Cat. No. / ID: 34362
Caractéristiques
- Marqueur His-tag exprimé optimisé pour l’élimination par l’enzyme TAGZyme
- Élimination performante du marqueur His-tag : > 95 % en seulement 30 minutes à 37 °C
- Expression de haut niveau des protéines marquées à l’histidine sur l’extrémité N-terminale
- Produits finis de grande pureté
- Élimination complète des contaminants par la méthode Ni-NTA
Détails produit
La TAGZyme DAPase Enzyme contient suffisamment d’enzyme pour une élimination hautement spécifique et performante du marqueur His-tag d’une quantité de protéine marquée à l’histidine allant jusqu’à 10 mg. Le TAGZyme System peut être utilisé pour l’élimination du marqueur His-tag de protéines contenant un point d’arrêt de DAPase intrinsèque exprimé avec le TAGZyme pQE-2 Vector.
Performances
La TAGZyme DAPase Enzyme élimine efficacement les dipeptides dans l’ordre, des marqueurs His-tag sur l’extrémité N-terminale jusqu’au « point d’arrêt » exprimé avec le TAGZyme pQE-2 Vector.
Principe
Les protéines recombinantes marquées à l’histidine sont devenues précieuses dans l’étude de la structure et du fonctionnement des protéines. Compte tenu de la petite taille et de la faible immunogénicité du marqueur His-tag, son élimination n’est pas toujours nécessaire. Pour autant, un produit de protéine dépourvu d’acides aminés dérivés de vecteurs est préférable pour certaines applications, comme les études de détermination de la structure par rayons X ou RMN, ou la production de traitements.
Le TAGZyme pQE-2 Vector est adapté aux protéines contenant un point d’arrêt de DAPase intrinsèque.
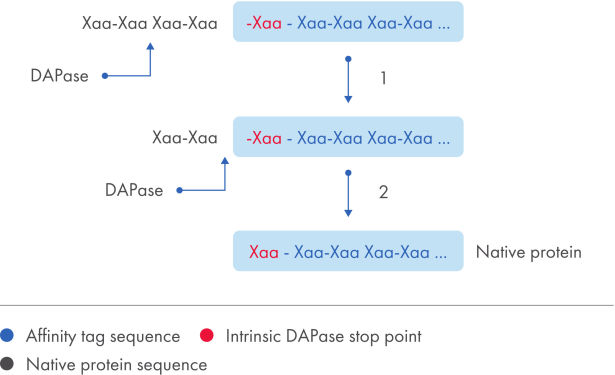
Le TAGZyme System élimine les marqueurs His-tag sur l’extrémité N-terminale des protéines recombinantes présentant une spécificité et une efficacité élevées. L’enzyme DAPase permet de cliver dans l’ordre les dipeptides à partir de l’extrémité N-terminale de la protéine marquée à l’histidine purifiée (consultez l’illustration « Élimination du marqueur His-tag »). La digestion est interrompue dès que l’enzyme atteint un « point d’arrêt », un motif d’acide aminé qui ne peut pas servir de substrat (consultez le tableau « Points d’arrêt de DAPase »).
Points d’arrêt de DAPase
| Acide aminé | Séquence de points d’arrêt de DAPase (↓)* |
|---|---|
| Lysine (Lys, K) | Xaa-Xaa...Xaa-Xaa ↓ Lys-Xaa... |
| Arginine (Arg, R) | Xaa-Xaa...Xaa-Xaa ↓ Arg-Xaa... |
| Proline (Pro, P) | Xaa-Xaa...Xaa-Xaa ↓ Xaa-Xaa-Pro-Xaa... |
| Proline (Pro, P) | Xaa-Xaa...Xaa-Xaa ↓ Xaa-Pro-Xaa-Xaa... |
| Glutamine (Gln, Q)† | Xaa-Xaa...Xaa-Xaa ↓ Gln-Xaa... |
| Isoleucine (Ile, I) | Xaa-Xaa...Xaa-Xaa ↓ Xaa-Ile-Xaa-Xaa... |
Voir les illustrations
Procédure
Grâce aux protéines recombinantes qui contiennent des points d’arrêt intrinsèques, l’expression avec le TAGZyme pQE-2 Vector permet une élimination complète et efficace du marqueur His-tag N-terminal, quel que soit le site de clonage de l’insert d’ADN (consultez l’illustration « Élimination du marqueur His-tag »). Après incubation avec l’enzyme DAPase, le mélange réactionnel est soumis à une chromatographie d’affinité sur ions métalliques immobilisés (IMAC) soustractive avec une matrice de Ni-NTA (consultez l’illustration « Purification de protéines non marquées »). Les fragments du marqueur His-tag et la TAGZyme DAPase Enzyme (qui porte un marqueur 6xHis-tag C-terminal) se lient à la matrice, puis une protéine pure, non marquée, est récupérée dans la fraction d’effluent.
Voir les illustrations
Applications
Le TAGZyme System permet un clivage spécifique, l’utilisation de réactifs recombinants et l’élimination complète de tous les contaminants, cela en fait une méthode de choix pour la production de protéines dépourvues de marqueur His-tag pour des applications telles que :
- Détermination de la structure des protéines par RMN ou cristallographie aux rayons X
- Production de protéines à visée thérapeutique
Données et illustrations utiles
Élimination du marqueur His-tag.
Synthèse schématisée de la stratégie globale de clivage avec des enzymes TAGZyme.Clivage avec enzyme DAPase d’un marqueur His-tag N-terminal à partir d’une protéine contenant un point d’arrêt naturel pour obtenir la protéine cible mature.