✓ Traitement automatique des commandes en ligne 24 h/24 7 j/7
✓ Assistance technique et produits pertinente et professionnelle
✓ Commande (ou réapprovisionnement) rapide et fiable
QIAamp DNA Blood Mini Kit (50)
N° de réf. / ID. 51104
✓ Traitement automatique des commandes en ligne 24 h/24 7 j/7
✓ Assistance technique et produits pertinente et professionnelle
✓ Commande (ou réapprovisionnement) rapide et fiable
Caractéristiques
- Purification rapide d’un ADN de haute qualité, prêt à l’emploi
- Pas d’extraction organique ni de précipitation à l’alcool
- Rendements importants et homogènes
- Élimination complète des contaminants et des inhibiteurs pour des résultats fiables
- Formats de kits pour bas et haut débit – options d’automatisation pour tous les kits
Détails produit
Les QIAamp DNA Blood Kits fournissent une purification de l’ADN à base de membranes de silice à partir de sang total, de plasma, de sérum et d’autres liquides corporels. Les kits sont conçus pour le traitement d’échantillons de sang total humain frais ou congelé de 200 μl à 10 ml. Les colonnes de centrifugation QIAamp peuvent être facilement traitées à l’aide d’une centrifugeuse ou d’une rampe à vide. Un format de 96 puits pratique utilisant la centrifugation permet une purification de l’ADN dans les laboratoires nécessitant un haut débit de purification d’ADN à partir de sang, de couche leucocytaire, de plasma, de sérum, de moelle osseuse, de lymphocytes et de liquides corporels. Un kit dédié est également disponible pour la purification automatisée de 1 à 12 échantillons sur le QIAcube Connect.
Performances
Les QIAamp DNA Blood Kits offrent un rendement d’ADN de taille comprise entre 200 pb et 50 kb, en fonction de l’âge et des conditions de conservation des échantillons (voir la figure « Bandes apoptotiques dans le sang conservé »). L’ADN purifié est adapté pour l’amplification PCR long-range (voir la figure « PCR long-range ») et l’analyse par polymorphisme de longueur des fragments de restriction (RFLP) utilisée, par exemple, pour les tests de paternité (voir la figure « Test de paternité par analyse RFPL »).
Le QIAamp DNA Blood Midi Kit permet une récupération d’ADN allant jusqu’à 94,5 % ; le QIAamp DNA Blood Maxi Kit permet une récupération d’ADN allant jusqu’à 95,8 %, en fonction de la densité cellulaire de départ (voir le tableau « Rendements d’ADN à partir de sang humain total avec des densités cellulaires différentes »).
| Leucocytes par ml | Rendement d’ADN (µg) | Récupération d’ADN (%) | ||
| QIAamp Midi | QIAamp Maxi | QIAamp Midi | QIAamp Maxi | |
| 2,5 x 105 | 3,0 | 15,8 | 92,3 | 95,8 |
| 1,0 x 106 | 11,0 | 60,4 | 91,7 | 91,5 |
| 5,0 x 106 | 62,4 | 312,0 | 94,5 | 94,5 |
| 1,0 x 107 | 116,3 | 624,6 | 88,1 | 94,6 |
L’ADN génomique a été purifié à partir de 2 ml (QIAamp Midi) ou de 10 ml (QIAamp Maxi) de sang humain total et élué dans un tampon d’élution de 300 μl (Midi) ou 1 ml (Maxi). Le premier éluat a été chargé dans la colonne une deuxième fois et de nouveau centrifugé (c.-à-d. réélué). Le pourcentage de récupération d’ADN a été calculé en partant du principe qu’un seul leucocyte contient 6,6 pg d’ADN.
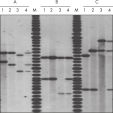
Le QIAamp 96 DNA Blood Kit traite les échantillons jusqu’à 200 µl, avec un temps de préparation de 192 échantillons de deux à trois heures. Il fournit un ADN particulièrement pur en moins d’une minute par préparation. Un débit encore plus élevé peut être obtenu en étalant la procédure. Les plaques QIAamp 96 offrent une uniformité sur l’ensemble des puits en matière de récupération d’ADN et de pureté (voir les figures « Reproductibilité des échantillons » et « Reproductibilité de rendement et de pureté »). Le rendement type est de l’ordre de 6 µg par 200 µl de sang total sain, avec un volume d’élution de 50 à 200 µl.
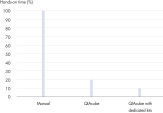
Le QIAamp DNA Blood Mini QIAcube Kit dédié permet l’isolement automatisé de l’ADN à partir de sang et l’isolement de l’ADN à partir de liquides corporels sur le QIAcube Connect. Le kit comprend des adaptateurs rotor préremplis avec des colonnes de centrifugation QIAamp et des tubes d’élution, ce qui procure un plus grand confort et un gain de temps significatif (voir la figure « Gain de temps significatif »). De plus, cela simplifie l’utilisation et minimise les erreurs utilisateur. Le contenu du kit dédié est conçu pour être utilisé avec la purification sur le QIAcube Connect, ce qui permet de réduire la quantité de déchets en éliminant les tubes superflus nécessaires à la procédure manuelle.
Si vous utilisez le QIAamp DNA Blood Mini Kit automatisable sur le QIAcube Connect, les QIAamp DNA Blood Mini Accessory Set A et QIAamp DNA Blood Mini Accessory Set B fournissent le confort de tampons et de réactifs supplémentaires pour l’automatisation de la préparation d’échantillons.
Principe
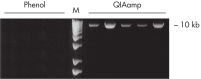
Aucune extraction au phénol-chloroforme n’est requise. L’ADN se lie spécifiquement à la membrane en gel de silice QIAamp alors que les contaminants la traversent. Les inhibiteurs de PCR, comme les cations divalents et les protéines, sont complètement éliminés au cours de deux étapes de lavage efficaces, laissant un acide nucléique pur à éluer dans de l’eau ou un tampon fourni dans le kit. La technologie QIAamp DNA Blood permet d’extraire un ADN génomique, mitochondrial ou viral à partir de sang et de liquides corporels associés, prêt à être utilisé en PCR et dans des procédures de transfert. La technologie de préparation des échantillons QIAamp est soumise à une licence.
Procédure
Les QIAamp DNA Blood Kits simplifient la purification de l’ADN à partir de sang et de liquides corporels grâce à des procédures rapides de centrifugation en colonnes, sous vide, de centrifugation ou automatisées (voir la figure « Procédure de la colonne de centrifugation QIAamp »). Du sang total frais et congelé avec des anticoagulants communs, tels que le citrate, l’EDTA ou l’héparine, peut être traité. Lors du traitement des colonnes de centrifugation QIAamp Midi ou Maxi à l’aide de rampes à vide, des Buffer AW1 [n° de réf. 19081] et Buffer AW2 [n° de réf. 19072] additionnels sont requis.
Traitement sous vide avec le QIAamp DNA Blood Mini Kit
Avec le QIAamp DNA Blood Mini Kit, il est possible de traiter le sang sous vide plutôt que par centrifugation pour une purification plus rapide et plus pratique de l’ADN. Les colonnes de centrifugation QIAamp Mini sont adaptées au collecteur QIAvac 24 à l’aide de VacValves et de VacConnectors. L’utilisation de VacValves est requise si les débits d’échantillon varient significativement, et ce, afin d’assurer un vide constant. Les VacConnectors à usage unique sont à utiliser pour éviter toute contamination croisée. L’utilisation des VacConnectors permet également d’effectuer ces procédures de centrifugation QIAamp sur le QIAvac 6S avec les QIAvac Luer Adapters.
Traitement automatisé sur le QIAcube Connect
Primé, le QIAcube Connect utilise une technologie de pointe pour le traitement des colonnes de centrifugation QIAGEN, qui permet une intégration harmonieuse de la préparation d’échantillons automatisée et à bas débit dans les flux de travail du laboratoire. Toutes les étapes de la procédure de purification sont entièrement automatisées ; jusqu’à 12 échantillons peuvent être traités par cycle d’exécution. Le QIAcube Connect, utilisé en combinaison avec le QIAamp DNA Mini QIAcube Kit dédié, est une solution idéale pour une purification de l’ADN rapide, facile et pratique.
Traitement haut débit avec le format de 96 puits
La procédure de centrifugation QIAamp 96 nécessite les profonds rotors libres du 96-Well-Plate Centrifugation System de QIAGEN pour s’adapter aux plaques QIAamp 96 empilées sur les blocs de 96 puits. Du sang total frais ou congelé traité avec des anticoagulants communs, tels que l’EDTA, le citrate ou l’héparine, peut être utilisé. Le sang total sec peut être traité avec un équipement supplémentaire et le protocole spécifique fourni par les services techniques de QIAGEN ou par votre distributeur local.
Applications
Les QIAamp DNA Blood Kits offrent la technologie éprouvée QIAamp pour la purification de l’ADN à partir d’une grande variété de matériaux. Sources d’échantillons :
- Sang total frais et congelé ou couche leucocytaire
- Plasma ou sérum
- Moelle osseuse
- Lymphocytes
- Plaquettes
- Liquides corporels
- Cellules de culture
- Écouvillons et cellules buccales
| Fonctionnalités | QIAamp DNA Blood Mini Kit | QIAamp DNA Blood Midi Kit | QIAamp DNA Blood Maxi Kit | QIAamp 96 DNA Blood Kit |
| Applications | PCR, PCR long-range, transfert de Southern | PCR, transfert de Southern | PCR, transfert | Transfert de Southern, cartographie génomique |
| Volume d’élution | 50 à 200 µl | 100 à 400 µl | 500 à 2 000 µl | 50 à 200 µl |
| Format | Colonne de centrifugation | Colonne de centrifugation | Colonne de centrifugation | Plaque de 96 puits |
| Type d’échantillon principal | Sang total, liquides corporels | Sang total, liquides corporels | Sang total, liquides corporels | Sang total, liquides corporels |
| Traitement | Manuel (centrifugation ou sous vide) | Manuel (centrifugation ou sous vide) | Manuel (centrifugation ou sous vide) | Manuel (centrifugation ou sous vide) |
| Purification de l’ARN total, du miARN, de l’ARNm poly A+, de l’ADN ou des protéines | ADN génomique, ADN mitochondrial, ADN viral | ADN génomique, ADN mitochondrial, ADN viral | ADN génomique, ADN mitochondrial, ADN viral | ADN génomique, ADN mitochondrial, ADN viral |
| Quantité d’échantillon | 1 à 200 µl | 0,3 à 2 ml | 3 à 10 ml | < 200 µl |
| Technologie | Technologie à base de silice | Technologie à base de silice | Technologie à base de silice | Technologie à base de silice |
| Durée par cycle d’exécution ou par préparation | 20 à 40 minutes | 55 minutes | 55 minutes | 2 à 3 heures (192 échantillons) |
| Rendement | 4 à 12 µg | 20 à 60 µg | 300 à 600 µg | 6 µg |
Données et illustrations utiles
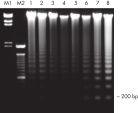
Répartition par taille type avec le QIAamp DNA Blood Mini Kit (sang)
Répartition par taille type d’ADN extrait de sang avec le QIAamp DNA Blood Mini Kit. Les échantillons ont été analysés avec le système Femto Pulse et les signaux ont été normalisés par rapport au pic maximal.