dPCR LNA Mutation Assay (200)
Kat.-Nr. / ID. 250200
| Popular Products | GeneGlobe ID |
|---|---|
| dPCR Mutation Assay ABL1 12573 Human | DMH0000573 |
| dPCR Mutation Assay ABL1 12574 Human | DMH0000053 |
| dPCR Mutation Assay WT1 27370 Human | DMH0000048 |
| dPCR Mutation Assay PIK3CA 12597 Human | DMH0000050 |
| dPCR Mutation Assay PIK3CA 776 Human | DMH0000062 |
Eigenschaften
- Das Duplex-Design des Assays detektiert mutierte und Wildtyp-Sequenzen
- Wahlweise FAM + HEX oder Atto 550 + ROX Fluoreszenzfarbstoff-Kombinationen
- LNA-angereicherte Primer und Sonden erhöhen die Spezifität und Sensitivität des Assays
- Im Nasslabor getestete dPCR-Assays für mehr als 200 Ziele verfügbar
- Geeignet für den Einsatz bei zirkulierender Tumor-DNA, Flüssigbiopsie, FFPE- und anderen Gewebeproben
- Zur Verwendung mit QIAcuity MasterMix
Angaben zum Produkt
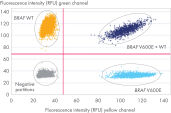
Die dPCR LNA Mutation Assays ermöglichen den Nachweis von individuellen Sequenzmutationen, die aus umfassenden kuratierten Datenbanken wie COSMIC ausgewählt wurden. Die Assay-Designs wurden im Labor mittels dPCR getestet, und die daraus resultierenden Daten liegen vor. Die Sensitivität für den Nachweis der Zielmutation beträgt 0,1 % bei einem Wildtyp-Hintergrund in einem Well einer QIAcuity Nanoplate. Eine noch höhere Sensitivität wird erreicht, wenn die Reaktion auf mehrere Wells aufgeteilt und die Auswertung kombiniert wird. Die Wahl zwischen zwei verschiedenen Fluoreszenzfarbstoffkombinationen ermöglicht den Nachweis von Mutanten- und Wildtypsequenzen sowie die Multiplex-Analyse von zwei Zielmutationen in einem Well.
Die Assays sind im Röhrchenformat als gebrauchsfertiger, 30-fach konzentrierter Assay erhältlich, der aus zwei Primern und Sonden für Mutante und Wildtyp besteht. dPCR LNA Mutation Assays sind für den Einsatz mit dem QIAcuity MasterMix vorgesehen.
Leistung
Die dPCR ermöglicht eine höhere Sensitivität beim Nachweis und bei der absoluten Quantifizierung von seltenen Ereignissen/Sequenzvarianten, sodass das Auftreten einer Mutation von 0,1 % vor einem Hintergrund aus genomischer Wildtyp-DNA detektiert werden kann. Die Partitionierung ermöglicht eine Erhöhung der spezifischen Konzentration des Ziels. Die Endpunkt-Amplifikation und Detektion oberhalb des Rauschens mit einem binären positiven/negativen Zielaufruf ermöglicht eine präzise Quantifizierung, die unabhängig von der Amplifikationsleistung ist (z. B. durch PCR-Inhibitoren).
Prinzip
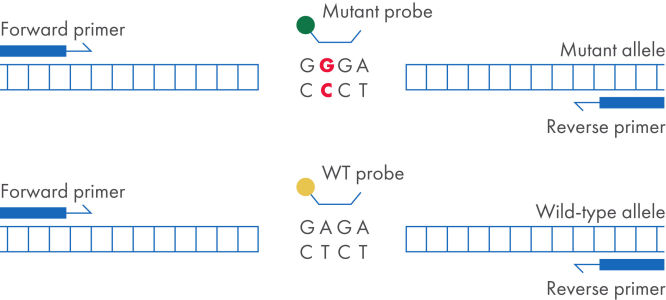
Die dPCR LNA Mutation Assays sind auf Hydrolysesonden basierende Duplex-Assays, die das Auftreten spezifischer Mutationssequenzen identifizieren. Eine Sonde detektiert das mutierte Allel, die andere Sonde das Wildtyp-Allel. Die digitale PCR ist das sensitivste und zuverlässigste Verfahren zum Nachweis einer mutierten DNA-Sequenz in einem Wildtyp-Hintergrund. Die dPCR LNA Mutation Assays nutzen LNA-verstärkte Primer und Sonden, die die Spezifität und Sensitivität des Assays erhöhen und den Nachweis einer Mutationsrate von 0,1 % in einem Hintergrund genomischer Wildtyp-DNA in einem einzigen Nanoplatten-Well ermöglichen.
Zwei Optionen für den Fluoreszenznachweis ermöglichen die Multiplex-Analyse von zwei Mutationszielen in einem Well. Bei der Produktkonfiguration kann zwischen FAM + HEX oder Atto 550 + ROX zum Nachweis von Mutante und Wildtyp gewählt werden.
Verfahren
Zur Durchführung des dPCR-Experiments geben Sie ein Aliquot der genomischen DNA-Probe (isoliert aus frischen, gefrorenen oder fixierten Proben) zu dem QIAcuity MasterMix und dem dPCR LNA Mutation PCR Assay hinzu. Jede Reaktion wird in einem oder mehreren Wells einer QIAcuity Nanoplate analysiert.
Nach dem Laden und Versiegeln der Nanoplatte wählen Sie das empfohlene Zyklusprogramm auf dem QIAcuity-Gerät. Bei der Analyseart der absoluten Quantifizierung gibt die QIAcuity Software Suite das Ergebnis als Kopien pro Mikroliter an. Unter dem Analysetyp Mutationsanalyse sind die berechnete fraktionierte Häufigkeit des mutierten Ziels und die entsprechenden Zahlenangaben aufgeführt.
Anwendungen
dPCR LNA Mutation PCR Assays eignen sich hervorragend für die rasche und genaue Identifizierung einzelner spezifischer DNA-Sequenzmutationen in frischen, gefrorenen oder fixierten Proben.
Ergänzende Daten und Abbildungen
dPCR LNA Mutation Assay-Konfiguration.