✓ 연중무휴 하루 24시간 자동 온라인 주문 처리
✓ 풍부한 지식과 전문성을 갖춘 제품 및 기술 지원
✓ 신속하고 안정적인 (재)주문
QIAamp DNA Blood Mini Kit (50)
카탈로그 번호 / ID. 51104
✓ 연중무휴 하루 24시간 자동 온라인 주문 처리
✓ 풍부한 지식과 전문성을 갖춘 제품 및 기술 지원
✓ 신속하고 안정적인 (재)주문
특징
- 즉시 사용 가능한 고품질 DNA의 신속한 정제
- 유기 추출 또는 알코올 침전물 없음
- 일관되고 높은 수율
- 신뢰할 수 있는 결과를 위한 오염 물질 및 억제제 완전 제거
- 저처리량부터 고처리량까지 다양한 키트 형식 – 모든 키트에 대한 자동화 옵션
제품 세부 정보
QIAamp DNA Blood Kit는 전혈, 혈장, 혈청 및 기타 체액에서 실리카 멤브레인으로 DNA를 정제합니다. 이 키트는 200μl부터 최대 10ml까지 다양한 샘플 크기의 신선 또는 냉동 인간 전혈 처리용으로 설계되었습니다. QIAamp 스핀 컬럼은 원심분리기나 진공 매니폴드에서 쉽게 다룰 수 있습니다. 원심분리를 이용한 편리한 96-well 포맷은 혈액, 연막, 혈장, 혈청, 골수, 림프구 및 체액에서 고처리량의 DNA를 정제해야 하는 실험실에서 DNA를 정제하는 데 적합합니다. 전용 키트는 QIAcube Connect에서 1~12개 검체를 자동으로 정제할 수 있습니다.
성능
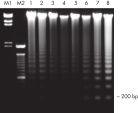
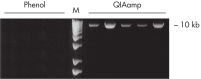
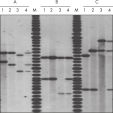
QIAamp DNA Blood Kit는 샘플의 보관 기간 및 보관 상태에 따라 200bp에서 50kb 까지의 다양한 크기의 DNA를 추출할 수 있습니다 (그림 '보관된 혈액의 세포사멸 밴드 패턴' 참조). 정제된 DNA는 긴 범위 PCR 증폭(long-range PCR amplification)(그림 ' 긴 범위 PCR' 참조) 및 제한효소 절편 길이 다형성(Restriction Fragment Length Polymorphism, RFLP) 분석, 예를 들면 친자 확인 검사(그림 ' RFLP 분석을 통한 친자 확인' 참조)에 적합합니다.
QIAamp DNA Blood Midi Kit는 최대 94.5%의 DNA 회수율을 보이고, QIAamp DNA Blood Maxi Kit는 시작 세포 밀도에 따라 최대 95.8%의 DNA 회수율을 보입니다(표 '세포 밀도 차이에 따른 인간 전혈의 DNA 수율' 참조).
| ml당 백혈구 | DNA 수율(µg) | DNA 회수율(%) | ||
| QIAamp Midi | QIAamp Maxi | QIAamp Midi | QIAamp Maxi | |
| 2.5 x 105 | 3.0 | 15.8 | 92.3 | 95.8 |
| 1.0 x 106 | 11.0 | 60.4 | 91.7 | 91.5 |
| 5.0 x 106 | 62.4 | 312.0 | 94.5 | 94.5 |
| 1.0 x 107 | 116.3 | 624.6 | 88.1 | 94.6 |
유전체 DNA는 2ml(QIAamp Midi) 또는 10ml(QIAamp Maxi)의 인간 전혈에서 정제하여 300µl(Midi) 또는 1ml(Maxi) 용출 완충액에서 용출했습니다. 첫 번째 용출액을 두 번째로 컬럼에 넣고 다시 원심분리했습니다(재용출). DNA 회수 백분율은 백혈구 하나당 6.6pg의 DNA를 함유하고 있다고 가정하여 계산했습니다.
QIAamp 96 DNA Blood Kit는 최대 200µl 크기의 샘플을 처리할 수 있으며, 2~3시간 안에 192개의 샘플을 준비하여 준비 회당 1분 이내에 고순도 DNA를 생산합니다. 시간차를 두고 절차를 진행하면 처리량을 더 높일 수 있습니다. QIAamp 96 플레이트는 웰 간 균일한 DNA 회수 및 순도를 제공합니다(그림 ' 샘플 재현성' 및 ' 수율 및 순도의 재현성' 참조). 일반적인 수율은 건강한 전혈 200μl당 6μg이며 용출량은 50~200μl입니다.
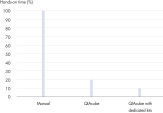
전용 QIAamp DNA Blood Mini QIAcube 키트를 사용하면 QIAcube Connect에서 혈액의 DNA를 자동으로 분리하고 체액에서 DNA를 분리할 수 있습니다. 이 키트에는 QIAamp 스핀 컬럼과 용출 튜브가 사전에 장착되어 있는 로터 어댑터가 포함되어 있어 편의성이 더욱 높으며 시간도 절약됩니다(그림 ' 상당한 시간 절약' 참조). 또한 사용 용이성이 향상되고 사용자 오류가 최소화됩니다. QIAcube Connect에서의 정제에 맞게 전용 키트의 내용물을 조정하였고 수동 절차에나 필요한 불필요한 튜브는 포함되지 않았기 때문에 낭비가 줄었습니다.
QIAcube Connect에서 자동화할 수 있는 QIAamp DNA Blood Mini Kit를 사용하는 경우 QIAamp DNA Blood Mini 액세서리 세트 A와 QIAamp DNA Blood Mini 액세서리 세트 B에서 자동화된 샘플 준비용으로 추가 완충액과 시약을 제공하므로 편리합니다.
원리
페놀-클로로포름 추출은 필요하지 않습니다. 오염 물질이 통과하는 동안 DNA는 QIAamp 실리카겔 멤브레인에 특이적으로 결합합니다. 2가 양이온 및 단백질과 같은 PCR 억제제는 효율적인 2단계 세척을 통해 완전히 제거되며, 키트와 함께 제공되는 정제수 또는 완충액에서 순수한 핵산이 용출됩니다. QIAamp DNA Blood 기술은 혈액 및 관련 체액에서 PCR 및 블로팅 절차에 바로 사용할 수 있는 유전체, 미토콘드리아 또는 바이러스 DNA를 생산합니다. QIAamp 샘플 준비 기술은 정식 라이선스를 받았습니다.
절차
QIAamp DNA Blood Kit는 신속 스핀 컬럼, 진공, 원심분리 또는 자동화 절차를 통해 혈액에서의 DNA 정제 및 체액에서의 DNA 정제를 간소화합니다(그림 ' QIAamp 스핀 컬럼 절차' 참조). 구연산염, EDTA, 헤파린과 같은 일반적인 항응고제가 첨가된 신선 또는 냉동 전혈을 처리할 수 있습니다. 진공 매니폴드에서 QIAamp Midi 또는 Maxi 스핀 컬럼을 처리하는 경우 진공 매니폴드와 함께 사용하려면 추가 버퍼 AW1[카탈로그 번호 19081] 및 버퍼 AW2[카탈로그 번호 19072]가 필요합니다.
QIAamp DNA Blood Mini Kit를 활용한 진공 처리 방식
QIAamp DNA Blood Mini Kit를 사용하면 원심분리 대신 진공 방식으로 혈액을 처리할 수 있어 DNA 정제 속도가 더욱 빨라지고 편리합니다. QIAamp mini 스핀 컬럼은 VacValves와 VacConnectors를 사용하는 QIAvac 24 매니폴드에 들어갑니다. 샘플 유량이 크게 다를 경우에 일관된 진공을 보장하려면 VacValve를 사용해야 합니다. 교차 오염을 방지하기 위해 일회용 VacConnector를 사용합니다. VacConnectors를 사용하면 QIAvac Luer 어댑터를 사용하는 QIAvac 6S에서도 이러한 QIAamp 스핀 절차를 수행할 수 있습니다.
QIAcube Connect에서의 자동화 처리
수상에 빛나는 QIAcube Connect는 첨단 기술을 사용하여 QIAGEN 스핀 컬럼을 처리하므로 자동화된 저처리량 샘플 준비를 실험실 워크플로에 원활하게 통합할 수 있습니다. 정제 절차의 모든 단계는 전면 자동화되어 있으며, 한 번 실행에 최대 12개의 샘플을 처리할 수 있습니다. QIAcube Connect를 전용 QIAamp DNA Mini QIAcube 키트와 함께 사용하는 조합은 DNA를 빠르고 쉽고 편리하게 정제할 수 있는 뛰어난 조합입니다.
96-well 형식의 고처리량 처리
QIAamp 96 스핀 절차에는 96-well 블록에 적재되는 QIAamp 96 플레이트를 수용하려면 QIAGEN 96-well 플레이트 원심분리 시스템의 딥 로터 버킷이 필요합니다. EDTA, 구연산염, 헤파린과 같은 일반적인 항응고제가 처리된 신선 또는 냉동 전혈을 사용할 수 있습니다. 건조 전혈은 QIAGEN 기술 서비스나 현지 유통업체에서 제공하는 추가 장비와 특수 프로토콜을 사용하여 처리할 수 있습니다.
응용 분야
QIAamp DNA Blood Kit는 다양한 물질에서 DNA를 정제하기 위한 입증된 QIAamp 기술을 제공합니다. 샘플 출처는 다음과 같습니다.
- 신선 및 냉동 전혈 또는 백혈구 연층
- 혈장 또는 혈청
- 골수
- 림프구
- 혈소판
- 체액
- 배양 세포
- 면봉 및 구강 세포
| 특징 | QIAamp DNA Blood Mini Kit | QIAamp DNA Blood Midi Kit | QIAamp DNA Blood Maxi Kit | QIAamp 96 DNA Blood Kit |
| 응용 분야 | PCR, 긴 범위 PCR(long-range PCR), 서던 블로팅 | PCR, 서던 블로팅 | PCR, 블로팅 | 서던 블로팅, 유전체 매핑 |
| 용출량 | 50~200µl | 100~400µl | 500~2000µl | 50~200µl |
| 형식 | 스핀 컬럼 | 스핀 컬럼 | 스핀 컬럼 | 96-well 플레이트 |
| 주 샘플 유형 | 전혈, 체액 | 전혈, 체액 | 전혈, 체액 | 전혈, 체액 |
| 처리 | 수동(원심분리 또는 진공) | 수동(원심분리 또는 진공) | 수동(원심분리 또는 진공) | 수동(원심분리 또는 진공) |
| 총RNA, miRNA, poly A+ mRNA, DNA 또는 단백질 정제 | 유전체 DNA, 미토콘드리아 DNA, 바이러스 DNA | 유전체 DNA, 미토콘드리아 DNA, 바이러스 DNA | 유전체 DNA, 미토콘드리아 DNA, 바이러스 DNA | 유전체 DNA, 미토콘드리아 DNA, 바이러스 DNA |
| 샘플양 | 1~200µl | 0.3~2ml | 3~10ml | <200µl |
| 기술 | 실리카 기술 | 실리카 기술 | 실리카 기술 | 실리카 기술 |
| 실행당 또는 준비당 시간 | 20~40분 | 55분 | 55분 | 2~3시간(192개 샘플) |
| 수율 | 4~12µg | 20~60µg | 300~600µg | 6µg |
지원되는 데이터 및 수치
QIAamp DNA Blood Mini Kit(혈액)를 사용한 크기 분포 예시
QIAamp DNA Blood Mini Kit를 사용하여 혈액에서 분리한 DNA 크기 분포 예시. 샘플은 Femto Pulse System을 사용하여 분석하고 신호는 최대 피크로 정규화했습니다.