✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
QIAamp DNA Blood Mini Kit(50)
カタログ番号 / ID. 51104
✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
特徴
- 高品質DNAの高速精製、すぐに使えるDNA
- 有機溶媒による抽出もアルコールの沈殿も不要
- 一貫性があり、高収量
- 汚染物質や阻害物質を完全に除去して高い信頼性の結果
- キットフォーマットは低スループットから高スループットまで対応可能、すべてのキットに自動化のオプション
製品詳細
QIAamp DNA Blood Kitは、シリカ膜を用いて全血、血漿、血清、その他の体液からDNAを精製するキットです。200 μlから最大10 mlの新鮮または冷凍されたヒト全血まで、さまざまなサンプル量に対応しています。QIAampスピンカラムは遠心分離機または真空マニホールドで簡単に処理でき、96ウェルフォーマットを用いた遠心分離で血液、バフィーコート、血漿、血清、骨髄、リンパ球、体液などからの高スループットなDNA精製が可能です。また、QIAcube Connectを使用し、1~12サンプルの自動精製を行う専用キットも提供しています。
パフォーマンス
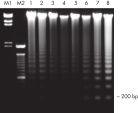
QIAamp DNA Blood Kitは、サンプルの経過時間と保存状態に応じて200 bp~50 kbの長さのDNAを回収できます(図 「保存血液におけるアポトーシス性結合」を参照)。精製されたDNAはロングレンジPCRによる増幅に最適で(図「 ロングレンジPCR」を参照)、制限断片長多型(RFLP)解析は、例えば父子鑑別に利用されます(図「 RFLP解析を使った父子鑑別」を参照)。
開始細胞の濃度に応じて、QIAamp DNA Blood Midi Kitは最大94.5%、QIAamp DNA Blood Maxi Kitは最大95.8%のDNAを回収可能です(表「異なる細胞濃度のヒト全血からのDNAの収率」)。
| 白血球/ml | DNA収量(µg) | DNA回収率(%) | ||
| QIAamp Midi | QIAamp Maxi | QIAamp Midi | QIAamp Maxi | |
| 2.5 x 105 | 3.0 | 15.8 | 92.3 | 95.8 |
| 1.0 x 106 | 11.0 | 60.4 | 91.7 | 91.5 |
| 5.0 x 106 | 62.4 | 312.0 | 94.5 | 94.5 |
| 1.0 x 107 | 116.3 | 624.6 | 88.1 | 94.6 |
DNA回収率は白血球1個が6.6 pgのDNAを含むという前提で算出されている。ゲノムDNAは、ヒト全血2 ml(QIAamp Midi)または10 ml(QIAamp Maxi)から精製され、300 µl(Midi)または1 ml(Maxi)の溶出バッファーで溶出されました。初回の溶出液は再度カラムにロードし、再度遠心分離を行いました(すなわち再溶出)。DNA回収率は、1個の白血球が6.6 pgのDNAを含むという前提で算出されました。
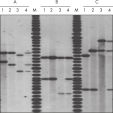
QIAamp 96 DNA Blood Kitは、最大200 µlのサンプルを処理でき、2~3時間で192サンプルを調製し、1回の処理につき1分未満で高純度のDNAを回収できます。手順をずらすことで、さらに高いスループットを実現することも可能です。QIAamp 96プレートは、DNA回収率と純度においてウェル間の均一性を提供します(図「 サンプルの再現性」および「 収量および純度の再現性」を参照)。通常の収量は200 µlの健康な全血から6 µgで、溶出量は50~200 µlです。
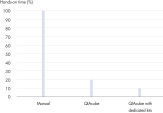
QIAcube Connectに専用のQIAamp DNA Blood Mini QIAcube Kitを使用すると、血液および体液からのDNA分離が自動化できます。キットには、あらかじめQIAampスピンカラムと溶出チューブが搭載されたローターアダプターが含まれており、時間と手間を大幅に節約できます(図「 時間と手間を大幅に節約」を参照)。さらに、使いやすさが向上し、ユーザーエラーの可能性を最小限にします。専用キットの内容物はQIAcube Connectを使った精製に合わせており、手動のプロセスに必要な余分なチューブは含まれていないため、廃棄物も削減されます。
自動化が可能なQIAamp DNA Blood Mini KitをQIAcube Connectで使用する場合、QIAamp DNA Blood Mini Accessory Set AおよびQIAamp DNA Blood Mini Accessory Set Bを併用すると、予備のバッファーと試薬が付属しており、自動でサンプル調製を行うのに便利です。
原理
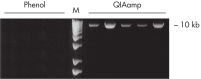
フェノール–クロロホルム抽出は必要ありません。DNAはQIAampシリカゲル膜に特異的に結合する一方、汚染物は通過します。2価カチオンやタンパク質などのPCR阻害物質は、2回の効率的な洗浄工程で完全に除去され、純粋な核酸のみが残り、水またはキット付属のバッファーに溶出されます。QIAamp Blood技術により、血液や関連する体液から、PCRやブロッティングのプロセスにすぐに使用できるゲノムDNA、ミトコンドリアDNA、ウイルスDNAを回収できます。QIAampサンプル調製技術は正式なライセンスを取得しています。
操作手順
QIAamp DNA Blood Kitは、血液からのDNA精製および体液からのDNA精製を、高速スピンカラム、真空プロトコール、遠心分離、または自動化されたプロセスを用いて簡略化します(「 QIAampスピンカラムのプロセス」を参照)。クエン酸塩、EDTA、ヘパリンなどの一般的な抗凝固剤を含む新鮮または冷凍された全血の処理も可能です。QIAamp MidiまたはMaxiスピンカラムを真空マニホールドで処理する場合、真空マニホールドで使用するために追加でBuffer AW1 [カタログ番号19081]およびBuffer AW2 [カタログ番号19072]が必要です。
QIAamp DNA Blood Mini Kitを使った真空プロトコール
QIAamp DNA Blood Mini Kitを使用すると、血液やその他の体液の処理が遠心分離ではなく真空プロトコールで行えるため、DNA精製の速度と利便性が大幅に向上します。QIAamp Miniスピンカラムは、QIAvac 24 PlusマニホールドにVacValveおよびVacConnectorを使用して収容されます。サンプルの流量が著しく異なる場合は、一貫した真空を確保するためにVacValvesを使用する必要があります。使い捨てのVacConnectorsは、クロスコンタミネーションを防ぐために使用します。また、VacConnectorsを使用することで、QIAvac Luerアダプターを搭載したQIAvac 6SでもQIAampスピン処理が可能になります。
QIAcube Connectを使った自動処理
アワードを受賞したQIAcube Connectは、QIAGENスピンカラムを用いた処理に最新技術を採用しており、自動化された低スループットのサンプル調製がラボのワークフローにシームレスに統合されます。精製プロセスのすべてのステップは完全に自動化され、1回のランで最大12サンプルを処理することができます。QIAcube Connectと専用のQIAamp DNA Mini QIAcube Kitを組み合わせることで、高速で簡単かつ利便性の高いDNA精製が実現します。
高スループットで96ウェルフォーマットを処理
QIAamp 96のスピンプロセスでは、96ウェルブロックの上にスタックしたQIAamp 96プレートを収納するために、QIAGEN 96-Well-Plate Centrifugation Systemの深型ローターバケットが必要です。使用できる血液は、EDTA、クエン酸塩、ヘパリンなどの一般的な抗凝固剤で処理された新鮮または冷凍の全血です。乾燥した全血は、追加の装置とQIAGENテクニカルサービスまたはお近くの販売店から提供される特別なプロトコールを用いて処理する必要があります。
アプリケーション
QIAamp DNA Blood Kitは、実績のあるQIAamp技術を活用し、さまざまな材料からDNAの精製を実現します。サンプルの種類には次のようなものがあります。
- 新鮮全血、冷凍全血、軟膜
- 血漿と血清
- 骨髄
- リンパ球
- 血小板
- 体液
- 培養細胞
- スワブと口腔細胞
| 特徴 | QIAamp DNA Blood Mini Kit | QIAamp DNA Blood Midi Kit | QIAamp DNA Blood Maxi Kit | QIAamp 96 DNA Blood Kit |
| アプリケーション | PCR、ロングレンジPCR、サザンブロッティング | PCR、サザンブロッティング | PCR、ブロッティング | サザンブロッティング、ゲノムマッピング |
| 溶出量 | 50~200 µl | 100~400 µl | 500~2000 µl | 50~200 µl |
| フォーマット | スピンカラム | スピンカラム | スピンカラム | 96ウェルプレート |
| 主なサンプルタイプ | 全血、体液 | 全血、体液 | 全血、体液 | 全血、体液 |
| 処理 | 手動(遠心分離または真空分離) | 手動(遠心分離または真空分離) | 手動(遠心分離または真空分離) | 手動(遠心分離または真空分離) |
| トータルRNA、miRNA、poly A+ mRNA、DNA、タンパク質の精製 | ゲノムDNA、ミトコンドリアDNA、ウイルスDNA | ゲノムDNA、ミトコンドリアDNA、ウイルスDNA | ゲノムDNA、ミトコンドリアDNA、ウイルスDNA | ゲノムDNA、ミトコンドリアDNA、ウイルスDNA |
| サンプル量 | 1~200 µl | 0.3~2 ml | 3~10 ml | 200 µl未満 |
| 技術 | シリカテクノロジー | シリカテクノロジー | シリカテクノロジー | シリカテクノロジー |
| ランあたり、または調製あたりの時間 | 20~40分 | 55分 | 55分 | 2~3時間(192 サンプル) |
| 収量 | 4~12 µg | 20~60 µg | 300~600 µg | 6 µg |
裏付けデータと数値
QIAamp DNA Blood Mini Kit(血液)を使用した際の典型的なDNAサイズ分布
QIAamp DNA Blood Mini Kitを使用して血液から得られたDNAの典型的なサイズ分布です。サンプルはFemto Pulse Systemで分析され、信号は最大ピークに基づいて正規化されました。