DNeasy Blood & Tissue Kit(50) icon_0368_ls_gen_eco_friendly-s
カタログ番号 / ID. 69504
特徴
- さまざまなサンプルタイプに使用できる標準化された方法
- 特殊サンプルでも高収量
- 高品質なDNA
- 出発物質の種類に応じた最適化プロトコール
- スピンカラムおよび96ウェル用高スループットフォーマット
- QIAcube Connectでワークフローを自動化する
製品詳細
DNeasy Blood & Tissue Kitsは、シリカベースのプロセスを用いて、フェノールやクロロホルムを使用せずに、スピンカラムおよび96ウェルプレートフォーマットで迅速にDNAを抽出します。ほとんどのサンプルにおいて、プロテイナーゼKを使用した直接的な溶解によって、機械的破砕の必要がなくなり、操作時間を短縮できます。特定のサンプルに合わせたプロトコールにより、高品質なDNAを安定して抽出できるため、ライフサイエンス、遺伝子型決定、獣医学的病原体の研究に最適です。また、キットはQIAcube Connectを使用することで自動化が可能です。
よりサステナブルな選択肢として、QIAwave DNA Blood & Tissue Kitは、プラスチックと段ボールの使用をそれぞれ最大62%と58%削減しています。キットに含まれる廃液チューブは100%再生プラスチック製で、手順全体で再利用できます。QIAwaveバッファーは濃縮されており、1ボトルあたりのプラスチック使用量を最大90%削減しています。外見に違いはありますが、QIAwave Kitは標準キットと同様に使いやすく、化学成分と性能は同一です。なお、バッファーの再調製には滅菌ガラス製ボトルが必要です。
My Green Labとの提携のもと、DNeasy Blood & Tissue Kit(50/250)およびQIAwave DNA Blood & Tissue Kit(50/250)の環境影響評価を実施しました。My Green LabのACT環境影響係数ラベルは、以下のようなさまざまなサステナビリティ基準に基づいて製品を評価し、ランク付けすることを目的としています。
- 製造
- 責任ある化学薬品管理
- サステナブルな製品の内容および包装材料
- 製品使用後のパッケージ処理
製品には1から10のスコアが付けられ、エネルギーと水の消費はそれぞれkWhやガロンごとに1点ずつ評価されます。低いスコアは、環境への負荷が低いことを意味します。詳細については、「DNeasy Blood & Tissue KitおよびQIAwave DNA Blood & Tissue Kit ACT環境影響係数ラベル」US( 50/ 250)、EU( 50/ 250)、UK( 50/ 250)」を参照してください。
パフォーマンス
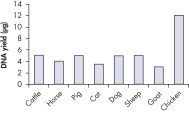
DNeasy DNA Blood & Tissue KitおよびQIAwave DNA Blood & Tissue Kitは効率が良く、動物血液や組織から全DNAを高収量で抽出します(表「動物組織からの一般的なDNA収量」 と図 DNAの収量を参照してください)。プロトコールが最適化され、例えば動物の毛のほか、培養細胞、固定組織、グラム陽性菌、グラム陰性菌といった非標準サンプルからも高い収量が確実に回収できます( ウマの遺伝子型決定を参照)。酵母、昆虫、毛髪などその他のサンプルタイプからのDNA精製用のプロトコールは専用オンラインから入手していただけます。
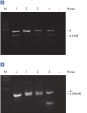
QIAwave DNA Blood & Tissue KitとDNeasy Blood & Tissue Kitは化学成分が同一であるため、性能も同じです。どちらも競合キットより優れていることが実証されています(図「QIAwave DNA Blood & Tissue Kitの性能」を参照)。
QIAwave DNA Blood & Tissue Kitの標準プロトコールに従うことで、動物血液および組織サンプルから高収量の全DNAを回収できます(表「QIAwave DNA Blood & Tissueを使用した動物組織からの典型的なDNA収量」と図「 DNA収量」を参照)。さらに、以下のような非標準サンプルからも高収量を得られるように最適化されたプロトコールもお求めいただけます。
- 獣毛
- 培養細胞
- グラム陽性菌、グラム陰性菌
- 酵母
- 昆虫
- その他のサンプルタイプ
| 由来 | 量 | DNA(µg) |
| 哺乳類の血液 | 100 µl | 3~6 |
| 鳥類の血液 | 5 µl | 9~40 |
| HeLa細胞 | 2 x 106 | 15~25 |
| 肝臓 | 25 mg | 10~30 |
| 脳 | 25 mg | 15~30 |
| 腎臓 | 25 mg | 15~30 |
| 脾臓 | 10 mg | 5~30 |
| マウス尾 | 1.2 cm(先端部) | 10~25 |
| マウス尾 | 0.6 cm(先端部) | 20~40 |
| ブタの耳 | 25 mg | 10~30 |
| ウマの毛 | 10本 | 2~4 |
| 魚のひれ | 20 mg | 10~20 |
| 魚卵(サバ) | 10 mg | 5~10 |
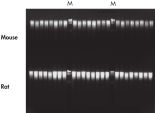
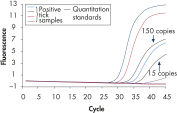
DNeasy Blood & Tissue Kitを使えば、ライフサイエンス、獣医学、遺伝子型決定のアプリケーションで一般的に使用される動物種を含むさまざまなサンプルタイプからDNAを精製する手順を簡略化できます( 高品質DNAを参照)。精製されたDNAには、PCR阻害物質が存在せず、標準なマルチプレックスPCR( 効果的な16plex PCRを参照)およびreal-time PCR( Real-time PCRを参照)を対象とした高感度検出が可能になります。DNeasy Blood & Tissue Kitは、遺伝子導入マウスの実験室での解析(高スループット精製を参照)から家畜の育種プログラム( ブタの血統遺伝子型決定を参照)まで、信頼性の高い結果が得られます。定期検査アプリケーションは、DNeasy 96 Blood & Tissue Kitを使用すれば簡単にスケールアップが図れます(高スループット精製を参照)。
原理
DNeasy and QIAwave DNA Blood & Tissue Kitは、新鮮または冷凍保存された動物組織や細胞、血液、細菌など、さまざまなサンプルソースから全DNA(例えば、ゲノムDNA、ミトコンドリアDNA、病原体DNA)を迅速に精製できるように設計されています。
DNeasy膜は、シリカベース膜の結合特性と、シンプルなマイクロスピン技術、またはQIAGEN 96ウェルプレート遠心分離システムを組み合わせたものです。DNAは、高濃度のカオトロピック塩が溶液中の水和分子から水分を除去する働きのもと、DNeasy膜に吸着します。DNeasy Blood & Tissueプロセスのバッファー条件は、DNAがシリカ膜に特異的に吸着し、汚染物質や酵素阻害物質を最適に除去できるように設計されています。
DNeasy Blood & Tissue Kitを使った精製は、フェノールやクロロホルムによる抽出やアルコール沈殿を必要とせず、最小限の操作で済むため、複数のサンプルを同時に処理するのに非常に適しています。より高スループットのアプリケーションには、DNeasy 96 Blood & Tissue Kitを使用することで、96または192のサンプルを同時に処理することが可能です。
操作手順
使いやすいスピンカラムまたは96ウェルフォーマットに搭載された高い信頼性のシリカ膜技術で、迅速で再現性の高いDNA精製を実現する一方、有機溶媒による抽出やアルコール沈殿が不要になります。(see DNeasy Miniおよび96のプロセスを参照)。サンプルを最初にプロテイナーゼKで溶解します。バッファーは最適なDNA結合条件を整えた状態になっています。溶解物をDNeasy Mini spin columnまたはDNeasy 96 plateにロードし、遠心分離すると、DNAは選択的に膜と結合しますが、汚染物質は通過します。残った汚染物質と酵素阻害物質は次に、2回にわたる効果的な洗浄工程で除去され、DNAが水またはバッファーに、すぐに使える状態で溶出されます。
DNeasy Blood & Tissue Kitに付属するDNeasy Miniスピンカラムはあらかじめ採取チューブの中に包装され、個別に密閉されているので、使いやすく安全です。DNeasy Miniスピンカラムを用いた精製プロセスはQIAcube Connectを使えば自動化が可能です。DNeasy 96 Blood & Tissue Kitは、QIAGEN 96-Well-Plate Centrifugation Systemを使った96ウェルフォーマットの精製で高いスループットを実現します。
DNeasy Blood & Tissue Kitに対応した標準プロトコールは、TRACKMAN Connectedシステムを使用することで、シームレスに実行できます。
アプリケーション
DNeasy Blood & Tissue KitとQIAwave DNA Blood & Tissue Kitを使うことで、以下のdPCRおよびNGS アプリケーションを含むすべてのダウンストリームアッセイにすぐに使える高品質DNAが抽出できます。
- ライフサイエンス研究
- 家畜の育種
- 血統遺伝子型決定
- 獣医学的病原体研究
- ルーチン適用検査
裏付けデータと数値
DNA Blood & Tissue Kits (50)ACT環境影響係数ラベルUS。
ACT環境影響係数ラベルは製品をいくつかのサステナビリティの基準から評価し、スコア付けできるように設計されています。製品には1から10のスコアが付けられますが、エネルギーと水の消費は、kWhかガロンごとに1点になります。低い点は、環境影響が低いことを意味します。QIAwave DNA Blood & Tissue Kit(50)は米国において同等の標準キットDNeasy Blood & Tissue Kit(50)と比べてEIFが21.5%低くなっています。

仕様
| 特徴 | 仕様 |
|---|---|
| Applications | PCR、real-time PCR、遺伝子型決定 |
| Elution volume | 100~200 µl |
| Time per run or per prep | 20分~1時間 |
| Main sample type | 血液、組織 |
| Format | 96ウェルプレート、スピンカラム |
| Sample amount | 100 µl/25 mg |
| Processing | 手動 |
| Yield | 6 µg/30 µg |
| Technology | シリカテクノロジー |
| Purification of total RNA, miRNA, poly A+ mRNA, DNA or protein | DNA |