✓ Automatische Verarbeitung von Online-Bestellungen 24/7
✓ Sachkundiger und professioneller technischer und Produkt-Support
✓ Schnelle und zuverlässige (Nach-)Bestellung
QIAamp DNA Blood Mini Kit (50)
Kat.-Nr. / ID. 51104
✓ Automatische Verarbeitung von Online-Bestellungen 24/7
✓ Sachkundiger und professioneller technischer und Produkt-Support
✓ Schnelle und zuverlässige (Nach-)Bestellung
Eigenschaften
- Schnelle Aufreinigung von hochwertiger gebrauchsfertiger DNA
- Keine organische Extraktion oder Alkoholausfällung
- Konsistent hohe Ausbeute
- Vollständige Entfernung von Verunreinigungen und Inhibitoren für zuverlässige Ergebnisse
- Kitformate für niedrigen bis hohen Durchsatz – Optionen für die Automatisierung aller Kits
Angaben zum Produkt
QIAamp DNA Blood Kits ermöglichen die Aufreinigung von DNA aus Vollblut, Plasma, Serum und anderen Körperflüssigkeiten auf der Basis von Silikamembranen. Die Kits sind für eine Bandbreite von Probengrößen von 200 μl bis zu 10 ml frischem oder gefrorenem menschlichem Vollblut ausgelegt. QIAamp-Spin-Säulen können problemlos in einer Zentrifuge oder auf Vakuumverteilern verarbeitet werden. Ein praktisches 96-Well-Format unter Verwendung von Zentrifugation ermöglicht die Aufreinigung von DNA für Labore, die einen hohen Durchsatz an DNA-Aufreinigung aus Blut, Buffy Coat, Plasma, Serum, Knochenmark, Lymphozyten und Körperflüssigkeiten benötigen. Für die automatische Aufreinigung von 1–12 Proben auf dem QIAcube Connect ist ebenfalls ein spezielles Kit erhältlich.
Leistung
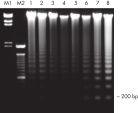
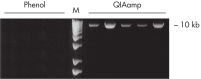
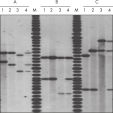
Die QIAamp DNA Blood Kits liefern DNA in einer Größe von 200 bp bis 50 kb, abhängig von Alter und Lagerung der Proben (siehe Abbildung „Apoptotische Bänderung in gelagertem Blut“). Die aufgereinigte DNA eignet sich für die Long-range-PCR-Amplifikation (siehe Abbildung „ Long-range-PCR“) und die Restriktionsfragmentlängen-Polymorphismus-Analyse (RFLP), die z. B. für Vaterschaftstests verwendet wird (siehe Abbildung „ Vaterschaftstests durch RFLP-Analyse“).
Das QIAamp DNA Blood Midi Kit liefert eine DNA-Rückgewinnungsrate von bis zu 94,5 %, das QIAamp DNA Blood Maxi Kit eine DNA-Rückgewinnungsrate von bis zu 95,8 %, abhängig von der Ausgangszelldichte (siehe Tabelle „DNA-Ausbeute aus menschlichem Vollblut bei unterschiedlichen Zelldichten“).
| Leukozyten pro ml | DNA-Ausbeute (µg) | DNA-Rückgewinnungsrate (%) | ||
| QIAamp Midi | QIAamp Maxi | QIAamp Midi | QIAamp Maxi | |
| 2,5 x 105 | 3,0 | 15,8 | 92,3 | 95,8 |
| 1,0 x 106 | 11,0 | 60,4 | 91,7 | 91,5 |
| 5,0 x 106 | 62,4 | 312,0 | 94,5 | 94,5 |
| 1,0 x 107 | 116,3 | 624,6 | 88,1 | 94,6 |
Genomische DNA wurde aus 2 ml (QIAamp Midi) oder 10 ml (QIAamp Maxi) menschlichem Vollblut aufgereinigt und in 300 µl (Midi) oder 1 ml (Maxi) Elutionspuffer eluiert. Das erste Eluat wurde ein zweites Mal auf die Säule gegeben und erneut zentrifugiert (d. h. erneut eluiert). Die prozentuale DNA-Rückgewinnungsrate wurde unter der Annahme berechnet, dass ein Leukozyt 6,6 pg DNA enthält.
Das QIAamp 96 DNA Blood Kit verarbeitet bis zu 200 µl Probenvolumen mit einer Präparationszeit von zwei bis drei Stunden für 192 Proben und liefert hochreine DNA in weniger als einer Minute pro Präparation. Ein noch höherer Durchsatz kann durch eine Staffelung des Verfahrens erreicht werden. QIAamp-96-Platten bieten eine einheitliche DNA-Ausbeute und -Reinheit von Well zu Well (siehe Abbildungen „ Probenreproduzierbarkeit“ und „ Reproduzierbarkeit von Ausbeute und Reinheit“). Die typische Ausbeute liegt bei 6 µg pro 200 µl gesundem Vollblut mit einem Elutionsvolumen von 50–200 µl.
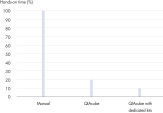
Das spezielle QIAamp DNA Blood Mini QIAcube Kit ermöglicht die automatisierte DNA-Isolierung aus Blut und aus Körperflüssigkeiten auf dem QIAcube Connect. Das Kit enthält Rotoradapter, die bereits mit QIAamp-Spin-Säulen und Elutionsröhrchen bestückt sind, was zu mehr Komfort und Zeitersparnis führt (siehe Abbildung „ Signifikante Zeitersparnis“). Darüber hinaus wird die Benutzerfreundlichkeit erhöht und Benutzerfehler werden minimiert. Der Abfall wird reduziert, da der Inhalt des speziellen Kits auf die Aufreinigung mit dem QIAcube Connect zugeschnitten ist und die überflüssigen Röhrchen, die für das manuelle Verfahren benötigt werden, nicht enthalten sind.
Wenn Sie das automatisierbare QIAamp DNA Blood Mini Kit auf dem QIAcube Connect verwenden, bieten das QIAamp DNA Blood Mini Accessory Set A und das QIAamp DNA Blood Mini Accessory Set B zusätzliche Puffer und Reagenzien für die automatisierte Probenpräparation.
Prinzip
Es ist keine Phenol-Chloroform-Extraktion erforderlich. Die DNA bindet spezifisch an die QIAamp-Silikagelmembran, während Verunreinigungen durchlaufen. PCR-Inhibitoren, z. B. zweiwertige Kationen und Proteine, werden in zwei effizienten Waschschritten vollständig entfernt, sodass reine Nukleinsäure entweder in Wasser oder in einem mit dem Kit gelieferten Puffer eluiert werden kann. Mit der QIAamp DNA Blood-Technologie wird genomische, mitochondriale oder virale DNA aus Blut und verwandten Körperflüssigkeiten gewonnen, die direkt einsatzbereit für PCR- und Blotting-Verfahren ist. Die QIAamp-Probenvorbereitungstechnologie ist vollständig lizenziert.
Verfahren
QIAamp DNA Blood Kits vereinfachen die DNA-Aufreinigung aus Blut und aus Körperflüssigkeiten mit schnellen Spin-Säulen-, Vakuum-, Zentrifugations- oder automatisierten Verfahren (siehe Abbildung „ QIAamp-Spin-Column-Verfahren“). Frisches und gefrorenes Vollblut mit gängigen Antikoagulantien wie Citrat, EDTA und Heparin kann verarbeitet werden. Bei der Verarbeitung von QIAamp Midi- oder Maxi-Spin-Säulen auf Vakuumverteilern werden zusätzlich Buffer AW1 [Kat.-Nr. 19081] und Buffer AW2 [Kat.-Nr. 19072] benötigt.
Vakuumprozess mit dem QIAamp DNA Blood Mini Kit
Mit dem QIAamp DNA Blood Mini Kit kann Blut durch Vakuum statt durch Zentrifugation verarbeitet werden, was eine schnellere und bequemere DNA-Aufreinigung ermöglicht. Die QIAamp Mini-Spin-Säulen werden mithilfe von VacValves und VacConnectors auf dem QIAvac 24-Vakuumverteiler angebracht. VacValves sollten verwendet werden, wenn die Durchflussraten der Proben erheblich voneinander abweichen, um ein gleichmäßiges Vakuum zu gewährleisten. Um Kreuzkontaminationen zu vermeiden, werden Einweg-VacConnectors verwendet. Mithilfe von VacConnectors können diese QIAamp-Spin-Verfahren auch auf QIAvac 6S mit QIAvac Luer Adaptern durchgeführt werden.
Automatisierte Verarbeitung auf dem QIAcube Connect
Der preisgekrönte QIAcube Connect nutzt eine fortschrittliche Technologie zur Verarbeitung von QIAGEN-Spin-Säulen und ermöglicht die nahtlose Integration von automatisierter Probenpräparation im geringen Durchsatz in die Laborabläufe. Alle Schritte des Aufreinigungsverfahrens sind vollständig automatisiert – bis zu 12 Proben können pro Lauf bearbeitet werden. Der QIAcube Connect bildet zusammen mit dem QIAamp DNA Mini QIAcube Kit eine erfolgreiche Kombination für eine schnelle, einfache und bequeme DNA-Aufreinigung.
Hochdurchsatzverarbeitung im 96-Well-Format
Beim QIAamp 96-Spin-Verfahren werden die tiefen Rotorbecher des QIAGEN 96-Well-Plate Centrifugation Systems benötigt, um die auf 96-Well-Blöcken gestapelten QIAamp 96-Platten aufzunehmen. Es kann frisches oder gefrorenes Vollblut verwendet werden, das mit gängigen Antikoagulantien wie EDTA, Citrat und Heparin behandelt wurde. Getrocknetes Vollblut kann mit zusätzlichen Geräten und unter Verwendung eines speziellen Protokolls verarbeitet werden, das beim Technischen Service von QIAGEN oder bei Ihrem örtlichen Händler erhältlich ist.
Anwendungen
QIAamp DNA Blood Kits bieten die bewährte QIAamp-Technologie für die Aufreinigung von DNA aus einer Vielzahl von Materialien. Beispiele für Probenquellen sind:
- Frisches und gefrorenes Vollblut oder Buffy Coat
- Plasma oder Serum
- Knochenmark
- Lymphozyten
- Blutplättchen
- Körperflüssigkeiten
- Zellkulturen
- Abstriche und Wangenzellen
| Eigenschaften | QIAamp DNA Blood Mini Kit | QIAamp DNA Blood Midi Kit | QIAamp DNA Blood Maxi Kit | QIAamp 96 DNA Blood Kit |
| Anwendungen | PCR, Long-range PCR, Southern Blotting | PCR, Southern Blotting | PCR, Blotting | Southern Blotting, Genome Mapping |
| Elutionsvolumen | 50–200 µl | 100–400 µl | 500–2000 µl | 50–200 µl |
| Format | Spin-Säule | Spin-Säule | Spin-Säule | 96-Well-Platte |
| Hauptprobentyp | Vollblut, Körperflüssigkeiten | Vollblut, Körperflüssigkeiten | Vollblut, Körperflüssigkeiten | Vollblut, Körperflüssigkeiten |
| Verfahren | Manuell (Zentrifugation oder Vakuum) | Manuell (Zentrifugation oder Vakuum) | Manuell (Zentrifugation oder Vakuum) | Manuell (Zentrifugation oder Vakuum) |
| Aufreinigung von Gesamt-RNA, miRNA, poly A+-mRNA, DNA oder Protein | Genomische DNA, mitochondriale DNA, virale DNA | Genomische DNA, mitochondriale DNA, virale DNA | Genomische DNA, mitochondriale DNA, virale DNA | Genomische DNA, mitochondriale DNA, virale DNA |
| Probenmenge | 1–200 µl | 0,3–2 ml | 3–10 ml | <200 µl |
| Technologie | Silika-Technologie | Silika-Technologie | Silika-Technologie | Silika-Technologie |
| Zeit pro Lauf oder Präparation | 20–40 Minuten | 55 Minuten | 55 Minuten | 2–3 Stunden (192 Proben) |
| Ausbeute | 4–12 µg | 20–60 µg | 300–600 µg | 6 µg |
Ergänzende Daten und Abbildungen
Exemplarische Größenverteilung mit dem QIAamp DNA Blood Mini Kit (Blut)
Beispielhafte Größenverteilung von DNA aus Blut mit dem QIAamp DNA Blood Mini Kit. Die Proben wurden mit dem Femto Pulse System analysiert und die Signale wurden auf den Maximalpeak normalisiert.