QIAamp DNA FFPE Advanced Kit (50)
Kat.-Nr. / ID. 56604
Eigenschaften
- Hohe Rückgewinnung von amplifizierbarer DNA
- Paraffinentfernung ohne Xylol oder ähnliche Lösungsmittel
- Uracil (desaminiertes Cytosin) – Schritt zur Entfernung von Artefakten unter Verwendung von Uracil-N-Glykosylase (UNG) während der DNA-Extraktion
- Gebrauchsfertige DNA für PCR, digitale PCR (dPCR) und Sequenzierung der nächsten Generation (NGS)
Angaben zum Produkt
Erhöhen Sie die Gewinnung hochwertiger DNA aus FFPE-Gewebe mit den QIAamp DNA FFPE Advanced Kits, die eine xylolfreie Entparaffinierung ohne Waschschritte, ein Doppellyseprotokoll und die UCP-Spin-Säulen-Technologie (aus ultrareiner Produktion) bieten.
Entfernen Sie C→U-Transitionen von Nukleinsäuren mit dem optionalen UNG-Verdau der Kits, um die gewonnene DNA für die NGS-Analyse zu optimieren.
Sie können die QIAamp DNA FFPE Advanced-Protokolle auch auf dem QIAcube Connect automatisieren.>
Leistung
Ein Merkmal der FFPE-DNA-Quantifizierung ist, dass verschiedene Methoden unterschiedliche Ergebnisse liefern. Hohe UV-VIS- oder fluorometrische Werte bedeuten dabei nicht unbedingt eine gute PCR-Leistung.
Da die Real-time PCR, oder quantitative PCR (qPCR), eine der häufigsten nachgelagerten Anwendungen für FFPE ist, sind die QIAamp DNA FFPE Advanced Kits für die qPCR optimiert.
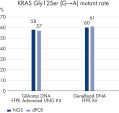
Unabhängig von den bei UV-VIS- oder fluorometrischen Messungen erzielten Werten zeigten die QIAamp DNA FFPE Advanced Kits bei der qPCR-Quantifizierung eine konsistent bessere PCR-Leistung als die anderen getesteten Produkte (siehe Abbildung „ Optimiert für PCR-Leistung“).
Das QIAamp DNA FFPE Advanced UNG Kit ist auch gut geeignet für die Aufreinigung von DNA, die für NGS-Analysen verwendet werden soll, da es das Problem von C→T/G→A-Transitionsartefakten löst, die in FFPE-Material aufgrund von Cytosin-Desaminierung häufig auftreten (siehe „ C→T/G→A-Transitionsartefakte“).
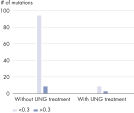
Durch den UNG-basierten Uracil-Verdau während der DNA-Isolierung reduziert das QIAamp DNA FFPE Advanced UNG-Protokoll falsch positive Meldungen von Einzel-Nukleotid-Varianten (single-nucleotide variants, SNVs) bei der NGS (siehe Abbildung „ Drastische Reduzierung von artefaktischen C→T | G→A-Transitionen“).
Prinzip
Die Präparation von DNA aus FFPE-Geweben birgt drei große Herausforderungen:
- Geringe Ausbeuten aufgrund von begrenztem Ausgangsmaterial und beeinträchtigter DNA
- Nicht amplifizierbare DNA aufgrund von formalininduzierten Quervernetzungen (Crosslinks)
- Artefakte durch desaminierte Cytosine können bei NGS für Mutationsanalysen zu falschen Ergebnissen führen
Die QIAamp DNA FFPE Advanced Kits maximieren DNA-Ausbeuten aus limitierten Probenmengen auf zweierlei Weise:
- Durch ein zweistufiges Lyseverfahren, das eine hohe DNA-Extraktion auch aus schwierig zu lysierenden Proben gewährleistet
- Indem die lösungsmittelbasierte Paraffinentfernung durch die Deparaffinization Solution ersetzt wird, die alle Waschschritte vor der initialen Lyse eliminiert und so das Risiko des Verlusts von knappem Probenmaterial minimiert
Die Entfernung von Quervernetzungen erhöht die Gewinnung amplifizierbarer DNA zusätzlich (siehe Abbildung „ Optimiert für PCR-Leistung“).
Durch die optionale UNG-Behandlung vor der zweiten Lyse werden Artefakte durch desaminierte Cytosine entfernt, wodurch sich die DNA besonders gut für NGS-Analysen eignet (siehe Tabelle „ Bereit für NGS“ sowie die Abbildungen „ Zuverlässige dPCR- und NGS-Ergebnisse“ und „ Drastische Verringerung von artefaktischen C→T | G→A-Transitionen“).
Verfahren
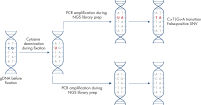
Das QIAamp DNA FFPE Advanced-Verfahren besteht aus einem vereinfachten Entparaffinierungsschritt, zwei Lyse-Schritten mit zwischengeschalteter Entfernung von Quervernetzungen und optionaler Artefaktentfernung sowie den Standardschritten Binden-Waschen-Eluieren (siehe Abbildung „ QIAamp DNA FFPE Advanced-Workflow“).
Anwendungen
DNA aus FFPE, die mit den QIAamp DNA FFPE Advanced Kits gewonnen wurde, kann sofort für PCR, dPCR oder NGS verwendet oder bei -30 °C bis -15 °C gelagert werden.
Ergänzende Daten und Abbildungen
QIAamp DNA FFPE Advanced-Workflow.
Das QIAamp DNA FFPE Advanced-Verfahren entfernt Paraffin ohne aufwendige wiederholte Waschschritte, indem Deparaffinization Solution anstelle von Xylol oder anderen Lösungsmitteln verwendet wird.
Die Lyse erfolgt mit Proteinase K. Vernetzungen werden durch 1-stündige Inkubation bei 90 °C entfernt. Artefakte können mit UNG entfernt werden.
Anschließend wird die RNA verdaut und die Probe ein zweites Mal lysiert, um die DNA-Gewinnung zu erhöhen.
Die DNA wird an die QIAamp UCP MinElute-Spin-Säule gebunden. Verunreinigungen werden mit Buffer AW1, Buffer AW2 und Ethanol weggewaschen, und die DNA wird in Buffer ATE eluiert.

Spezifikationen
| Eigenschaften | Spezifikationen |
|---|---|
| Applications | PCR, digitale PCR, Next-generation sequencing |
| Elution volume | 20–100 µl |
| Format | Spin-Säule |
| Main sample type | Formalin-fixierte, in Paraffin eingebettete (FFPE) Gewebeproben |
| Processing | Manuell oder automatisiert mit dem QIAcube Connect |
| Purification of total RNA, miRNA, poly A+ mRNA, DNA or protein | Genomische DNA |
| Sample amount | Gewebeschnitte, jeweils mit einer Dicke von 5–10 µm, mit einem Gesamtvolumen von 4 mm3 |
| Technology | Silika-Technologie |