✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
Cat. No. / ID: 206243
✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
特徴
- マルチプレックスPCRによる確実なマイクロサテライト解析
- 至適化なしにマイクロサテライトアッセイを確立
- 全フラグメントを良好かつ特異的に同時増幅
- 至適化済みプロトコールで正確な結果を迅速に実現
- 各種解析用装置を使用する場合の簡単なインストラクション
製品詳細
特異性の高いHotStarTaq Plus DNA Polymerase と特許を持つバッファーシステムをベースにしたType-it Microsatellite PCR Kitは、マルチプレックスPCRによるマイクロサテライト解析を確実に行ない、至適化は不要です。マスターミックスの全成分とその特殊な組み合わせにより、すべての遺伝子座を高い特異性で同時に増幅できます。本キットはマルチプレックスPCR を用いた迅速で信頼できるマイクロサテライト解析専用キットです。至適化済みのプロトコールには、高解像度キャピラリーシークエンシングあるいは電気泳動装置による解析についても記載されています。
パフォーマンス
図参照
原理
Type-it Microsatellite PCR Kitは、マイクロサテライト、STRあるいはVNTRマーカーを用いてヒト、動物、植物、バクテリアの迅速かつ正確なジェノタイピング解析を実現し、時間のかかる至適化実験は不要です。Type-it Microsatellite PCR Kitは、独自のQIAGENマルチプレックステクノロジーをベースにし、化学的修飾した特異性の高いホットスタート酵素、HotStarTaq Plus DNA Polymeraseおよびマルチプレックス用に特別に開発したFactor MPを含むバッファーシステムを含んでいます。これらの成分により高い収量が得られ、目的のマイクロサテライト遺伝子座がすべて確実に同時増幅できます。本キットは、マイクロサテライトまたはSTRの増幅のための専用プロトコルを含み、テンプレート量、サイクル数、およびPCR条件に関するステップごとのアドバイスと同様に、アガロースゲル、キャピラリーシークエンサー、Agilent Bioanalyzer、QIAxcel Advanced Systemなどのダウンストリーム解析のプラットフォーム用ごとに機器の詳細が含まれています。
Type-it Microsatellite PCR Buffer
画期的なType-it Microsatellite PCR Bufferにより複数のPCR増幅産物の増幅が容易になります。Type-it Microsatellite PCR Buffer は、特別に開発された塩と添加物のバランスの取れた組み合わせにより、従来のPCR試薬に比べ、反応中の全プライマーのアニーリングとエクステンションを同等の効率で増幅できます。このバッファーはユニークな配合比のKClと (NH4)2SO4を含み、従来のPCRバッファーに比べ、幅広い温度やMg2+濃度の範囲で厳密で特異的なプライマー・アニーリングを実現します。異なるアニーリング温度あるいはMg2+ 濃度を用いて行なうPCRの至適化は最小限ですみ、または不要なこともあります。従来のマルチプレックスPCR操作で行なう至適化操作は不要です。バッファーに入っている合成 Factor MP (図“ 安定で効果的なアニーリングの促進”)は、プライマー配列に関係なくプライマーのアニーリングとエクステンションを効率的に行ないます。Factor MPは、DNAテンプレート付近のプライマーの濃度を上げ、特異的に結合したプライマーを安定化します。
Q-Solution
Type-it Microsatellite PCR KitにはQ-Solutionが含まれています。この革新的なPCR 添加剤はDNAの変性環境を改善することにより、増幅困難なテンプレートの増幅を可能にします。このユニークな試薬を使用すると、PCR条件が最適でない場合でもPCR が可能になり、改良されることがあります。DMSOや他の他のPCR 添加物とは異なり、Q-Solution はどのプライマー・テンプレートシステムでも一定のワーキング濃度で使用し、毒性はありません。
図参照
操作手順
ルーチン解析のための信頼性の高い結果を保証するだけでなく、新しいアッセイの確立のために、高分解能キャピラリーシークエンサーでのマイクロサテライト解析用に最適化されたアプリケーション特有のプロトコールがType-it Microsatellite PCR Kitに含まれています。反応は室温で設定することができるので、使いやすく便利です。
迅速かつ簡単な方法で信頼できるジェノタイピング結果を実現
Type-it Microsatellite PCR Kit は、マルチプレックス・アッセイを迅速かつ簡単に確立でき、正確で再現性のあるジェノタイピング結果が得られます。マイクロサテライト、STR、SSRやVNTR解析など、どのようなアプリケーションでも、テンプレートとプライマーを添加し、至適化済みプロトコールに従ってサーマルサイクラープログラムをスタートするだけで実験ができます。反応ミックスには、マイクロサテライトに特異的なマルチプレックスPCRに必要な試薬が全て入っており、本キットを用いると、通常の方法のように時間がかかる至適化操作なしに正確な結果が得られます(図“ マルチプレックスPCRによる良好なジェノタイピング解析”)。
キャピラリーシークエンサーを用いた解析に至適化済み
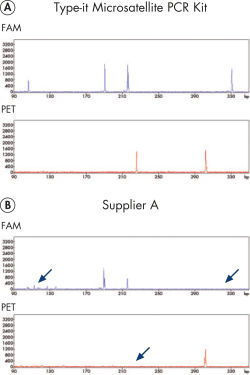
キャピラリーシークエンサーのような高分離能検出システムを用いたマイクロサテライト解析では、不均一な増幅産物量や蛍光シグナル強度が非常に異なるという問題が頻繁におこります。Type-it Microsatellite PCR Kit はこのような限界を克服し、マルチプレックス実験で全ての産物を高い収量で増幅できます。本キットは蛍光標識プライマーを使用する場合の至適化済みプロトコールが入っており、他社の酵素製品より性能が優れています(図“ 至適化不要の正確な 13-plex STR 解析”)。
図参照
アプリケーション
Type-it Microsatellite PCR Kitはマイクロサテライト、STRs(short tandem repeats)、SSRs(simple sequence repeats)、VNTRs(variable number of tandem repeats)を解析するために使用し、以下のような様々な研究分野で最適です:
- 生物種や個人の同定
- 血縁関係の解析
- マイクロサテライトの不安定性
- 集団遺伝学
裏付けデータと数値
Reliable 13-plex STR analysis without the need for optimization.
Specifications
| Features | Specifications |
|---|---|
| Applications | Multiplex PCR, Microsatellites, STRs, VNTRs, SSRs |
| Reaction type | PCR amplification |
| Product use | Functionally validated and developed for reliable Microsatellite Analysis |
| Mastermix | Yes |
| Real-time or endpoint | Endpoint |
| Sample/target type | Genomic DNA |
| Single or multiplex | Multiplex |
| With/without hotstart | With |




