✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
Cat. No. / ID: 206544
✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
特徴
- わずかな配列の違いも正確に検出
- 特異性の高いPCRにより増幅困難な遺伝子座を増幅
- 新しいEvaGreen 蛍光色素により明確な融解曲線
- 新規のHRMジェノタイピング解析実験系の設定を簡便化
- 簡便なマスターミックスフォーマットと至適化済みのプロトコール
製品詳細
Type-it HRM PCR Kit はHRM 解析により迅速で正確なジェノタイピングのための信頼できる製品です。本キットは増幅困難なゲノム遺伝子座でさえも良好に解析できるように至適化されています。本キットは、変異のスキャン用に最適であり、未知の変異のスクリーニングに使用できます。効率的に新しいHRMジェノタイピングアッセイを確立します。時間のかかるPCR実験条件の至適化なしに、非特異的な増幅を抑え、特異的な増幅産物および信頼できる結果が一定して得られます。従って、標準化や柔軟性を確保できるだけではなく、時間および経費の削減にも繋がります。本キットはRotor-Gene Q、Rotor-Gene 6000、LightCycler 480、Applied Biosystems 7500 Fast SystemのようなHRM 解析用に適したリアルタイムPCR装置において使用できます。
パフォーマンス
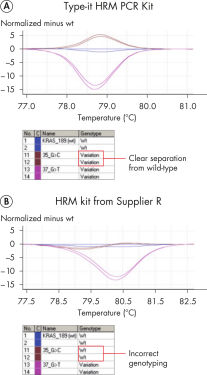
Type-it HRM PCR Kit は配列の違いを正確に解析できることが検証されており、画期的なHRMテクノロジーを用いてアレルを明確に識別するのに最適なキットです。HRMテクノロジーを用いた、高度なジェノタイピングアプリケーションなどで見られる未知で複雑な配列変異を迅速かつ簡便に検出・識別する最適な解析法です (図 " 高精度なジェノタイピング")。
わずかな配列の違いも正確に検出
本キットに付属のマスターミックスは、斬新な二本鎖DNA結合蛍光色素であるEvaGreenを含有し、至適化済みHRM buffer、HotStarTaq Plus DNA Polymerase、Q-Solution、dNTPsを含みます。そして、このような構成試薬はPCRの特異性を確実にし、解析困難な遺伝子座でも信頼できる結果を実現します。Type-it HRM PCR Kit は、テストを行なった他社のキットよりも優れており、非常に解析困難な微妙な配列の違いでさえも増幅が可能です。他の市販HRMマスターミックス試薬と比較して、Type-it HRM PCR Buffer のユニークな成分は、非常に厳密で特異的なプライマー結合を実現します(図 " 増幅困難な遺伝子の特異的で良好な増幅を実現")。他社のキットとは異なり、Type-it HRM PCR Kit を用いた場合、テストした全てのリアルタイムPCR装置で最初の実験から一定した性能を提供します。また、使用装置の性能によりClass IV SNPs(図 " A/T Class IV SNP の正確なジェノタイピング")や遺伝子変異(図 " 遺伝子変異の正確なタイピング")でさえも高精度な検出を実現します。これらの利点はまた再テストが必要なサンプル数を最小限に抑えることになり、時間と経費の節約にも繋がります。
迅速で簡便な変異スキャニング
Type-it HRM PCR Kitは信頼性の高い変異スキャニングを実現します。未知の遺伝子変異(insertions and deletions)を容易に検出し確実に区別でき、明確な融解曲線が得られます(図 " 正確な変異スキャニング")。
図参照
原理
HRM(High Resolution Melt) 解析とは、極めて精度の高い温度分解能を利用した融解曲線解析により、わずかなTm値のずれでさえも検出することができる技術です。温度の上昇に伴う二本鎖DNA(dsDNA) から一本鎖DNA(ssDNA)への解離(融解)挙動に基づいてPCR産物の解析を行ないます。PCR 産物は、その塩基配列、長さ、GC 含量に依存した融解温度を持ち、わずか1 塩基の違いまで判別できます。難しいジェノタイピングアプリケーションに見られるような未知の配列配列変異や複雑な配列変異でも、容易に解析できます(図 " 高精度なジェノタイピング")。さらに、HRMはプローブを利用したジェノタイピングに比べてより容易で経済的な方法であり、従来の方法とは違ってPCR産物のキャリーオーバー コンタミネーションを防ぎます。
ユニークなキット較正により、特異度の高い増幅が確実にできます(表)。
新しいEvaGreen 蛍光色素により明確な融解曲線
Type-it HRM PCR Kit は、二本鎖DNAに選択的に結合し、飽和できる第三世代の蛍光色素EvaGreen を含んでいます。SYBR® Green I に比べてEvaGreenはPCR阻害がないため高濃度で使用でき、GCリッチおよびATリッチ領域での結合アフィニティーは同等で、配列による優先性はありません。これらの特性があらゆるタイプのPCR産物のHRM解析にEvaGreenが最適な理由であり、わずかな蛍光強度の違いを可視化することで明確な融解曲線を作成でき、標準化された結果を実現します。
至適化済みのHRM PCR Master Mix
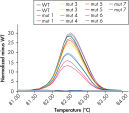
2x HRM PCR Master Mix はHotStarTaq Plus DNA Polymerase と画期的なHRM PCRバッファーシステムで構成され、Class IV SNPのような増幅困難な遺伝子座でも特異性の高い増幅と明確な融解曲線が得られます (図" A/T Class IV SNP の正確なジェノタイピング")。その結果、目的のPCR産物が高い特異性で確実に増幅され、非特異的増幅産物やプライマーダイマーの形成は最小限に抑えられます(" 遺伝子変異の正確なタイピング")。
画期的なバッファー(マスターミックスに含まれています)は、各PCRステップのアニーリングステップ中でプライマーの特異的結合の割合を非特異的結合に対して高めることにより、各PCRサイクルの特異的な増幅を実現します。本製品のバッファーは、独自のバランスでKClと(NH4)2SO4を含んでおり、従来のPCRバッファーよりも幅広い範囲のアニーリング温度とMg2+ 濃度条件で厳密なプライマーアニーリングが可能になります。最適なアニーリング温度やMg2+濃度を決定するためのPCRの至適化実験は、不要あるいは最小限に抑えられます。
増幅困難な変異遺伝子座に最適
Type-it HRM PCR Kit は増幅困難な変異遺伝子座を容易に検出する強力なシステムです (図 " 増幅困難な遺伝子の特異的で良好な増幅を実現")。GCリッチな領域あるいは高度な二次構造を有する領域に存在するSNPの変異は、従来のHRM解析用PCR製品では増幅が困難です。これらの問題を解決するために、2x HRM PCR Master Mix には画期的なPCR添加剤であるQ-Solution が特定の濃度で含まれています。Q-SolutionはDNAの変性環境を改善することによりPCR を改良し、至適化なしに増幅困難なターゲット配列の増幅を実現します。
| 構成 | 利点 |
|---|---|
| 2x HRM PCR Master Mix | 新しいアッセイの開発の際に最適化の必要がない 変異やSNPのHRM解析のために特別に開発された HRM解析に適した全サイクラーでの使用に専用のマスターミックス 反応セットアップが簡便で、ピペッティング誤差を最小にする |
| HotStarTaq Plus DNA Polymerase | 特異度の高い増幅 室温で迅速かつ簡単に反応セットアップ |
| Type-it HRM PCR Buffer | 明確な融解曲線 増幅の特異性が向上 |
| Q-Solution | 難しいテンプレートの増幅を改善 |
| EvaGreen Dye | 二本鎖DNA に飽和して結合する新しい色素 明確な融解曲線解析 |
図参照
操作手順
Type-it HRM PCR Kit には専用のアプリケーション別プロトコールが付属しており、Rotor-Gene Q、Rotor-Gene 6000、LightCycler 480をはじめとする様々なリアルタイムサイクラーで使用するためにあらかじめ最適化されています。さらに、キットに付属の至適化済みプロトコールを用いてApplied Biosystems 7500 Fast System および Applied Biosystems 7900 も使用可能です。詳細なプロトコールは、弊社のWebサイトで入手できます。
高速サイクリング操作と設定が簡便なHRM解析実験系により確実な結果
Type-it HRM PCR Buffer のユニークな性質とキットに添付の至適化済みのプロトコールにより、最初の実験から良好なHRMジェノタイピング結果が得られます。時間のかかる反応パラメーターの至適化は不要で、新しいHRMジェノタイピング解析実験系を迅速かつ容易に標準化でき、ルーチンの研究に組み込むことができます(図 " 至適化の必要なしに良好なHRMジェノタイピング解析")。さらに、Type-it HRM PCR Kit の高速サイクリングプロトコールはPCR反応時間を短縮することによりスループットを増大し、正確な結果を迅速に得られます。
簡便なキットフォーマット
本キットにはすでに至適化され即使用可能なマスターミックスフォーマットでお届けするので、さらに使いやすくなっています。マスターミックスを使用すると、ピペッティング操作は最小限に抑えられ、時間のかかる計算は不要なため、時間の節約、反応セットアップの簡略化、ピペッティングエラーやコンタミの発生源排除による再現性向上を実現します。マスターミックスを用いることで、迅速かつ簡単に室温で反応セットアップが行なえます。マスターミックスに含まれているHotStarTaq Plus DNA Polymeraseは、95℃、5分間のインキュベーションステップで活性化され、このステップは既存のサーマルサイクリングのプログラムに容易に導入できます。
図参照
アプリケーション
Type-it HRM PCR Kit は次のようなアプリケーションに使用可能です。
- 欠失、挿入、転座の検出
- 遺伝子変異のスキャニング
- SNPジェノタイピング
- 微生物変異体の検出と識別
Type-it HRM PCR Kit は以下のような研究分野において適応可能な一般的なツールです。
- 疾患および癌遺伝子座のタイピング
- バイオマーカーの探索
- 疾病関連遺伝子研究
- トランスジェニック植物/動物のタイピング
- 病原体検出とジェノタイピング
裏付けデータと数値
Successful typing of gene mutations.
Specifications
| Features | Specifications |
|---|---|
| Applications | Detection of SNPs, mutations and mutation scanning |
| Product use | Functionally validated and developed for reliable detection of genetic differences using HRM |
| Reaction type | PCR amplification |
| Enzyme activity | 5'-> 3' Exonuclease activity |
| With or without ROX | Without ROX |
| Sequence specific Probe | Not necessary, EvaGreen dye for detection included in the Mastermix |
| Sample/target type | Genomic DNA |
| With/without hotstart | With |
| Mastermix | Yes |
| Real-time or endpoint | Both |