Type-it Fast SNP Probe PCR Kit
TaqMan またはTaqMan MGBプローブを用いた正確で信頼できるSNP ジェノタイピング
TaqMan またはTaqMan MGBプローブを用いた正確で信頼できるSNP ジェノタイピング
✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
Cat. No. / ID: 206045
✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
特異性の高いHotStarTaq Plus DNA Polymerase と新しく開発されたバッファーシステムをベースにしたType-it Fast SNP Probe PCR Kitは、信頼できる明確なアレル識別を実現します。マスターミックス中の全成分の組み合わせにより、正確で特異性の高いプローブ結合を実現します。TaqMan SNP Genotyping Assays により検証されたType-it Fast SNP Probe PCR Kit は、増幅の難しいGC リッチなSNP 遺伝子座や微量のスタートテンプレートでも再現性のあるSNPジェノタイピングを実現します。
Type-it Fast SNP Probe PCR Kitは一定して高精度なSNPジェノタイピングを実現します。
Type-it Fast SNP Genotyping PCR Master Mix は特異性の高い HotStarTaq Plus DNA Polymerase と新しく開発されたSNPジェノタイピングPCRバッファーシステムで構成されているため、特異的なプローブ結合および一定した強い蛍光シグナルを実現します。市販されている他社のSNPジェノタイピング用マスターミックス試薬と比べて、アレルのクラスターがより明確に分離できます(図“ 特異性の高いプローブ結合”)。
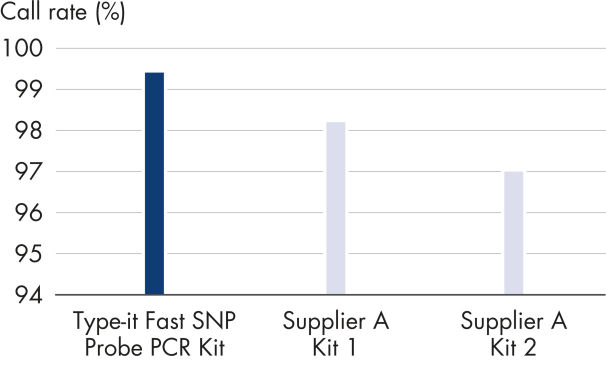
Type-it Fast SNP Probe PCR Kit は、各アレルのクラスターを明確に分離し、同一アレルがまとまったクラスターを形成するので、信頼できるSNP ジェノタイピングが可能で、その結果タイピングが困難なターゲットやSNP遺伝子座でも高いコールレートが得られます(図“ 自動アレルコーリングで最高の成功率”)。他社のSNP ジェノタイピング用キットと比較して、Type-it Fast SNP Probe PCR Kitは、1 ng のテンプレートDNA からでも、密集したクラスタリングを実現します(図“ 微量テンプレートからでも信頼できるSNPジェノタイピング”)。
Type-it Fast SNP Probe PCR Kitは簡便なマスターミックス・フォーマットで、特異性の高いHotStarTaq Plus DNA Polymeraseおよび迅速で効率的な増幅を実現するためにデザインされたSNPジェノタイピング用バッファーシステムで構成されています。この画期的なキットはアレルのばらつきのないクラスタリングおよび明確な分離を実現し、高いコールレートおよび正確で再現性の高い確実な結果が得られます(表)。
HotStarTaq Plus DNA Polymeraseはマスターミックス中に含まれ、少量のテンプレートでも特異性の高い増幅を実現します。 HotStarTaq Plus DNA Polymeraseは、常温では不活性状態でポリメラーゼ活性はありません。この特性により低温でのミスプライムによる増幅産物やプライマーダイマーの形成を抑制できます。
マスターミックス成分であるType-it Fast SNP PCR Buffer はまた、配列特異的な 5'-ヌクレアーゼプローブを用いた高速SNPジェノタイピング用に特化されています。Type-it Fast SNP PCR Buffer のユニークな成分により高い厳密性と特異性でアレルに特異的なプローブの結合が可能です (match probe)。プローブの融解挙動を変えることにより、融解温度のウィンドを狭めます。オリジナルQIAGEN PCR Bufferをベースにしたこの革新的なバッファーは、各PCRサイクルの短いアニーリングステップ中で特異的プライマー結合の非特異的プライマー結合への比率を高めます。このPCRバッファーはユニークな配合比のKClと (NH4)2SO4を含み、従来のPCRバッファーに比べ、幅広いアニーリング温度やMg2+濃度の範囲で厳密で特異的なプライマー・アニーリングを実現します。従って、異なるアニーリング温度あるいはMg2+ 濃度を用いて行なうPCRの至適化は最小限ですみ、または不要なこともあります。
Type-it Fast SNP PCR buffer に添加されているQ-Solutionは増幅困難なゲノム領域やSNP遺伝子座の反応条件を提供します。斬新なQ-BondテクノロジーによりSNPジェノタイピング結果が迅速に得られます(図“ アニーリング中の高速サイクリングのメカニズム”)。Type-it Fast SNP Probe PCR Kit のマスターミクス中のROX濃度はまたApplied Biosystems社の全装置でのSNP ジェノタイピングに対応できます。しかしROX色素はパッシブリファレンスとしてROXが不要なその他の装置でも使用可能です(表)。
| キット成分 | 特長 |
|---|---|
| 2x Master Mixフォーマット* | TaqMan MGBプローブによるSNPジェノタイピングのために開発 SNPジェノタイピングを全てのサイクラーで使用できるように最適化 |
| HotStarTaq Plus DNA Polymerase | 室温で迅速かつ簡単に反応セットアップ可能 少量のテンプレートでも特異性の高い増幅 |
| Type-it Fast SNP Probe PCR Buffer | 各アレルのクラスターを明確に分離 アレルのまとまったクラスタおよび高いコールレート プローブの結合特異性を増大 Q-Bond Moleculeによる高速サイクリング |
| Q-Solution | GCリッチのSNP遺伝子座でもスキャッタープロット解析でまとまったクラスターと強度なシグナル 最適でないアレルコーリングをさらに改善可能 |
Type-it Fast SNP Probe PCR Kitは市販のSNPジェノタイピングアッセイにより性能を実証済みで、TaqMan MGBプローブや、TaqMan MGB、TaqMan、その他ダブル標識プローブを用いてユーザーが開発したプローブベースのアッセイにも使用可能です。キットに付属の至適化済みのプロトコールにより、迅速かつ確実な解析を行なえます。
Type-it Fast SNP Probe PCR Master Mixは、配列特異的なプローブを用いた高速サイクリングPCRに適した反応条件を実現します。 本バッファーは独自のQ-Bond Moleculeを含み、標準的なサーマルサイクラーだけでなく、高速な加熱・冷却機能を持つサーマルサイクラーでもサイクリング時間を短縮します。高速サイクリング法はPCRラン時間を最高40%まで短縮することによりサンプル処理数を増大できます(“ ジェノタイピングのワークフロー”)。
本キット にはすでに至適化され即使用可能なマスターミックスが添付され、さらに使いやすくなっています。マスターミックスを使用すると、時間の節約、反応セットアップの簡略化、ピペッティングエラーやコンタミの発生源排除による再現性向上を実現します — ピペッティング操作は最小限に抑えられ、時間のかかる計算は不要です。HotStarTaq Plus DNA Polymerase(マスターミックスに含有) は、95℃、5分間のインキュベーションステップで活性化され、このステップは既存のサーマルサイクリングのプログラムに容易に導入できます。マスターミックスを用いて、反応セットアップを室温で迅速かつ容易に行なえます。
Type-it Fast SNP Probe PCR Kit は、蛍光強度プレートリード解析で使用するリアルタイムPCR装置にフィットするoptical PCR plate をセット可能な高速温度変化対応サイクラーでも、標準のサイクラーでもSNP PCRアッセイの増幅に至適化されています(表参照)。
| サイクラー | モデル |
|---|---|
| リアルタイムPCRサイクラー | QIAGEN:Rotor-Gene Q ABI PRISM 7900 (all series) Applied Biosystems 7500 (all series) Applied Biosystems 7300 ABI PRISM 7700 ABI PRISM 7000 ABI StepOne and StepOnePlus Bio-Rad: iCycler iQ Roche:LightCycler 480 Stratagene:Mx3000P およびMx3005P |
| スタンダードなサイクラ― | 標準的なランプ速度のサイクラー (例; GeneAmp 9700) 高速ランプ速度のサイクラー (例;GeneAmp 9800) |
Type-it Fast SNP Probe PCR Kit はTaqMan MGBプローブを用いたSNP検出や以下のような様々な分野で使用可能です:
Average automated allele call rates for a panel of different DNAs were analyzed using 6 different TaqMan® MGB-based SNP genotyping assays and 1 ng of genomic DNA from each of the 60 samples. PCR was performed with the indicated products in 384-well plates in a 5 μl reaction volume. Allelic discrimination plate read was performed on an Applied Biosystems 7900HT instrument. The Type-it Fast SNP Probe PCR Kit consistently resulted in the highest call rates and the lowest error rates.
| Features | Specifications |
|---|---|
| Applications | Probe-based SNP Genotyping |
| Product use | Functionally validated and developed for reliable SNP Genotyping |
| Mastermix | Yes |
| Real-time or endpoint | Both |
| With or without ROX | ROX included in Master Mix |
| With/without hotstart | With |
| Sample/target type | Genomic DNA |
| Enzyme activity | 5'-> 3' Exonuclease activity |
| Sequence specific Probe | TaqMan® Genotyping Assays, TaqMan® or TaqMan® MGB probes |
| Reaction type | PCR amplification |