miRCURY LNA miRNA PCR Assay
Kat.-Nr. / ID. 339306
| Popular Products | GeneGlobe ID |
|---|---|
| hsa-miR-10398-3p miRCURY LNA miRNA PCR Assay | YP02129529 |
| hsa-miR-510-3p miRCURY LNA miRNA PCR Assay | YP02127499 |
| hsa-miR-12119 miRCURY LNA miRNA PCR Assay | YP02126954 |
| hsa-miR-34a-3p miRCURY LNA miRNA PCR Assay | YP00206061 |
| hsa-miR-520h miRCURY LNA miRNA PCR Assay | YP00206050 |
Eigenschaften
- Unübertroffene Sensitivität erlaubt miRNA-Quantifizierung ausgehend von nur 1 pg Gesamt-RNA
- Sehr gut geeignet für Proben mit geringer RNA-Ausbeute, wie Serum/Plasma, FFPE-Schnitte und LMD-Proben
- Hohe Spezifität unterscheidet eng verwandte miRNAs und reife miRNA von Vorläufern
- Schnelles und einfaches zweistufiges Protokoll in weniger als 3 Stunden
- Vollständige miRBase-Abdeckung ermöglicht miRNA-Profiling aller Organismen
Angaben zum Produkt
miRCURY LNA miRNA PCR Assays sind individuelle miRNA-PCR-Primersets, die eine hoch sensitive und spezifische miRNA-Quantifizierung mit dem miRCURY LNA miRNA PCR System erlauben. Sowohl die Vorwärts- als auch die Rückwärtsprimer für die PCR-Amplifikation sind miRNA-spezifisch und wurden mit der LNA-Technologie optimiert. Assays sind im Röhrchen- oder Plattenformat mit 200 Reaktionen pro Röhrchen bzw. Well erhältlich.
Sie benötigen ein Angebot für Ihr Forschungsprojekt oder möchten Ihr Projekt mit unserem Expertenteam besprechen? Kontaktieren Sie uns!
Leistung
Unübertroffene Sensitivität
Die außergewöhnliche Sensitivität der miRCURY LNA miRNA PCR Assays wird durch die Kombination von universeller reverser Transkription mit LNA-optimierten und Tm-normalisierten Primern erzielt. Diese Kombination ermöglicht die genaue und zuverlässige Quantifizierung der einzelnen miRNAs schon ab 1 pg Gesamt-RNA in der initialen cDNA-Erststrangsynthese (siehe Abbildung Genaue Quantifizierung schon ab einem Ausgangsmaterial von 1 pg Gesamt-RNA).
Verglichen mit anderen miRNA Real-time-PCR-Systemen, die entweder Primer mit Haarnadelstruktur oder Standard-DNA-Primer verwenden, liefern die LNA-optimierten Primer eine signifikant höhere Sensitivität, insbesondere bei AT-reichen miRNAs (siehe Abbildung LNA-optimierte Primer ermöglichen eine wesentliche höhere Sensitivität als DNA-Primer).
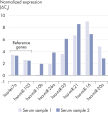
Die geringen Anforderungen an die Proben ermöglichen außerdem die miRNA-Quantifizierung bei Verwendung von Gesamt-RNA, die aus schwierigen Proben aufgereinigt wurde, wie etwa LMD-Proben und Serum/Plasma (siehe Abbildung Nachweis differentieller Expression von miRNAs in LMD-Proben aus FFPE-Gewebeschnitten and Unterschiede in der miRNA-Expression in verschiedenen Serumproben).
Vollständig validiert und optimiert
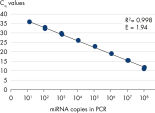
Alle Labor-validierten miRCURY LNA miRNA PCR Assays wurden auf die höchstmögliche Sensitivität optimiert. Über 80 % der Assays weisen mindestens 5 miRNA-Kopien in der PCR-Reaktion nach (siehe Abbildung Verdünnungsreihe von hsa-miR-181a). Die Primersets wurden auch für die spezifische Amplifikation des Ziels und für ein minimales Hintergrundsignal validiert (siehe Abbildung Spezifische und sensitive Amplifikation von miRNA).
Die einzige Plattform mit perfekter Spezifität
Die miRCURY LNA miRNA PCR Assays sind die einzige miRNA-Quantifizierungsplattform mit absoluter Spezifität (siehe die in Nature Methods veröffentlichte miRQC-Studie). In Spezifitätstest übertrafen sie eine andere sondenbasierte miRNA qPCR-Plattform (siehe Abbildung Spezifitätstest mit sondenbasiertem miRNA qPCR-System). Die perfekte Spezifität schließt falsch-positive Ergebnisse aus und gewährleistet nur robuste und zuverlässige miRNA-Signale.
Übelegene Diskriminierung
Die Einbeziehung von LNA in sowohl die Vorwärts- als auch die Rückwärtsprimer für die PCR-Amplifikation ermöglicht das Design von Assays, die zwischen miRNA-Sequenzen unterscheiden können, die um nur ein Nukleotid voneinander abweichen (siehe Abbildung Diskriminierung bis auf ein Nukleotid). Außerdem können die Assays zwischen reifen miRNA-Sequenzen und Vorläufer-miRNA unterscheiden.
Schnell, einfach und reproduzierbar
Das benutzerfreundliche, 3-stündige Protokoll spart Ihnen Zeit und Aufwand im Labor. In allen nachfolgenden PCR-Reaktionen wird die gleiche RT-Reaktion wie im Template verwendet, sodass das Verfahren im Vergleich zu Systemen, die eine miRNA-spezifische Erststrangsynthese erfordern, wesentlich vereinfacht wird. Die Anzahl der Pipettierschritte und damit die technische Variation werden auf ein Minimum reduziert. Dadurch wird es möglich, von Tag zu Tag und Standort zu Standort eine extrem hohe Reproduzierbarkeit zu erzielen.
miRCURY LNA miRNA PCR Assays wurden für die Verwendung mit dem miRCURY LNA RT Kit und dem miRCURY LNA SYBR Green PCR Kit optimiert. Die Verwendung anderer Reagenzien beeinflusst die Ergebnisse.
Prinzip
Ein einzigartiges System für das miRNA-Profiling
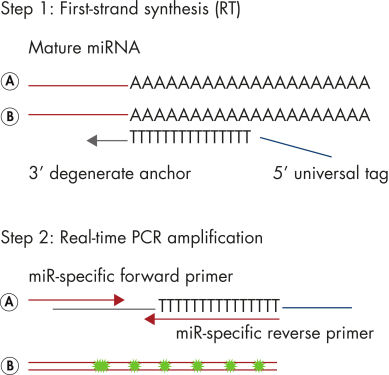
Die miRCURY LNA miRNA PCR Assays bieten durch Kombination aus universeller RT mit LNA-PCR-Amplifikation die beste auf dem microRNA Real-time-PCR-Markt verfügbare Kombination aus Leistung und Benutzerfreundlichkeit (siehe Abbildung Schematischer Überblick über das miRCURY LNA miRNA PCR-System). Die universelle RT ermöglicht die Verwendung einer cDNA-Erststrangsynthesereaktion als Template für mehrere miRNA-Real-time PCR-Assays. Dies spart wertvolle Probe sowie Zeit im Labor und reduziert technische Abweichungen. Darüber hinaus sind sowohl die Vorwärts- als auch die Rückwärtsprimer für die PCR-Amplifikation miRNA-spezifisch und wurden mittels LNA optimiert. Dadurch werden 1) eine außergewöhnliche Sensitivität und ein äußerst schwacher Hintergrund für eine genaue Quantifizierung sehr niedriger miRNA-Konzentrationen und 2) hochspezifische Assays, die die Unterscheidung zwischen eng verwandten miRNA-Sequenzen ermöglichen, erreicht.
Abdeckung
Es sind mehr als 20.000 Assays verfügbar, die alle Organismen in der miRBase 20 abdecken. Über 1.400 Assays wurden im Labor anhand von sowohl synthetischen als auch verschiedenen biologischen Proben vollständig im Hinblick auf Sensitivität, Spezifität, Effizienz und Hintergrund validiert. Die übrigen Assays wurden in silico anhand eines umfassenden Design-Algorithmus validiert, der hochqualitative, speziesspezifische und LNA-optimierte Assays mit optimaler Sensitivität und Spezifität für jeden Organismus gewährleistet. Das bedeutet, dass mehrere verschiedene Assays auf die gleiche Sequenz abzielen können. Letztendlich wird der Assay für jede Spezies basierend auf dem genetischen Hintergrund des Organismus ausgewählt. Falls Sie mit neuartigen miRNAs, z. B. aus einem NGS-Experiment, arbeiten, sind auch maßgeschneiderte LNA-miRNA-Primersets für jede beliebige miRNA verfügbar.
Referenzgen-Assays für die Normalisierung
Es sind neun Referenzgen-Primersets verfügbar. Sie ermöglichen eine qualitativ hochwertige Datennormalisierung und die Generierung zuverlässiger Daten aus vielen verschiedenen Probentypen (siehe nachstehende Tabelle). Diese Kontrollprimersets zielen auf endogene, kleine, nicht codierende RNAs ab, die konstitutiv und relativ stabil über verschiedene Gewebe hinweg exprimiert werden (siehe Abbildung Expressionsniveaus von Referenzgenen in verschiedenen humanen Geweben). Die Kontroll-Referenzgene decken einen großen Bereich von Cq-Werten ab und bieten die Möglichkeit zur genauen und zuverlässigen Normalisierung über ein breites Spektrum an miRNA-Expressionsniveaus.
Die Kontrollprimersets wurden als Referenzgene für das miRCURY LNA miRNA PCR System validiert und sind optimal für die Verwendung mit dem miRCURY LNA RT Kit und den miRCURY LNA SYBR Green PCR Kits geeignet. Es ist sehr wichtig, stets die konsistente und gleichbleibende Expression eines Gens in allen Geweben und bei allen Prozeduren innerhalb eines Experiments zu bestätigen, bevor ein Gen als Referenz gewählt wird. Wir empfehlen Ihnen, eine Reihe von Referenzgenen zu testen und für die Auswahl und Validierung von Referenzgenen eine Software wie GeNorm oder NormFinder (Teil der GenEx-Software) zu verwenden.
| Produktbezeichnung | Zielorganismus | Alternativnamen | NCBI-Referenz |
|---|---|---|---|
| Control primer set, SNORD38B (hsa) | hsa | U38B; RNU38B | NR_001457 |
| Control primer set, SNORD44 (hsa) | hsa | U44; RNU44 | NR_002750 |
| Control primer set, SNORD48 (hsa) | hsa | U48; RNU48 | NR_002745 |
| Control primer set, SNORD49A (hsa) | hsa | U49; U49A; RNU49 | NR_002744 |
| Control primer set, SNORA66 (hsa) | hsa | U66; RNU66 | NR_002444 |
| Control primer set, 5SrRNA (hsa) | hsa, mmu | V00589 | |
| Control primer set, U6-snRNA (hsa, mmu) | hsa, mmu, rno | U6 | X59362 |
| Control primer set, RNU5G snRNA (mmu, hsa) | mmu, hsa, rno | Rnu5a; U5a; Rnu5g | NR_002852 |
| Control primer set, RNU1A1 (mmu, hsa) | mmu, hsa, rno | Rnu1a-1; Rnu1a1 | NR_004411 |
| Control primer set, SNORD65 (mmu) | mmu | U65; RNU65 | NR_028541 |
| Control primer set, SNORD68 (mmu) | mmu | U68; RNU68 | NR_028128 |
| Control primer set, SNORD110 (mmu) | mmu | U110; RNU110 | NR_028547 |
Verfahren
Anwendungen
- Quantifizierung reifer miRNA
- snoRNA-Nachweis
Ergänzende Daten und Abbildungen
Schematischer Überblick über das miRCURY LNA miRNA PCR System